Перейти к:
Использование метода профилирования на основе коротких тандемных повторов для подтверждения подлинности клеточных линий в биобанках
https://doi.org/10.15829/1728-8800-2022-3386
Аннотация
Цель. Апробирование набора COrDIS (Гордиз, Россия) для подтверждения подлинности клеточных линий Биоресурсной коллекции ФГБУ "НМИЦ онкологии им. Н. Н. Блохина" Минздрава России методом профилирования на основе коротких тандемных повторов.
Материал и методы. Выбранный метод зарекомендовал себя в качестве надежного и воспроизводимого варианта. При таком подходе ряд полиморфных коротких тандемных повторов STR (short tandem repeat)-локусов амплифицируется с использованием коммерчески доступных наборов праймеров. Продукты полимеразной цепной реакции (ПЦР) анализируются одновременно со стандартами размера с использованием автоматических методов флуоресцентного обнаружения. Результаты предоставляются в виде простого числового кода, соответствующего длинам продуктов ПЦР, амплифицированных в каждом локусе. Применяя этот метод к клеточным линиям, лаборатория может, как проверить подлинность коммерческих клеточных линий, так и сформировать базу данных своих линий. В работе использовали набор отечественного производителя COrDIS "ЭКСПЕРТ 26" (Гордиз, Россия), валидированный для молекулярно-генетической идентификации личности на основе мультиплексного ПЦР-анализа 26-ти высоко-полиморфных локусов геномной дезоксирибонуклеиновой кислоты человека. Анализ результатов ПЦР проводили методом капиллярного электрофореза с использованием автоматического генетического анализатора с лазер-индуцированной флуоресцентной детекцией (Applied Biosystems 3500xL).
Результаты. При апробации метода проведено профилирование 37 клеточных линий, из которых 18 анонсированы в международных базах данных и 19 уникальных, полученных в ФГБУ "НМИЦ онкологии им. Н. Н. Блохина" Минздрава России, а также смеси клеточных линий с целью определения пределов выявления контаминации. Полученные результаты показали соответствие профилей коммерческих клеточных линий с данными в международных базах. В рамках данной работы были получены профили уникальных линий и заложено начало собственной генетической базы биоресурсной коллекции. Исследования на выявление предела обнаружения контаминации другой линией показали, что при содержании в общем пуле даже 4% культуры-контаминанта удается выявить ее отдельные аллели.
Заключение. Полученные результаты свидетельствуют о возможности использования метода для идентификации образцов коллекции и обнаружения внутривидовой контаминации.
Ключевые слова
Для цитирования:
Кособокова Е.Н., Мальченкова А.А., Калинина Н.А., Косоруков В.С. Использование метода профилирования на основе коротких тандемных повторов для подтверждения подлинности клеточных линий в биобанках. Кардиоваскулярная терапия и профилактика. 2022;21(11):3386. https://doi.org/10.15829/1728-8800-2022-3386
For citation:
Kosobokova E.N., Malchenkova A.A., Kalinina N.A., Kosorukov V.S. Using short tandem repeat profiling to validate cell lines in biobanks. Cardiovascular Therapy and Prevention. 2022;21(11):3386. (In Russ.) https://doi.org/10.15829/1728-8800-2022-3386
Введение
Широкий спектр исследований, проводимых на клеточных линиях, включая изучение механизмов действия, эффективности, безопасности новых терапевтических препаратов, механизмов лекарственной резистентности опухолей и многие другие аспекты, сделал этот объект чрезвычайно значимым в современной науке и технологии.
Значительная часть исследований, представленных в международных литературных источниках, подвергается сомнению в связи с отсутствием подтверждения подлинности и чистоты использованной клеточной линии [1-3], в то время как контаминация другой линией, а также вирусная и микоплазменная контаминации являются частыми1 и общеизвестными проблемами, широко обсуждаемыми в научном сообществе [4][5].
Контроль подлинности используемой в исследовании клеточной линии до сих пор не стал общепринятым, несмотря на широко известные в научном сообществе проблемы, связанные с работами, проведенными на ошибочно идентифицированных культурах, приведшими к распространению дезинформации в научных источниках и обладающими сомнительной ценностью [6]. Результатом пренебрежения процедурами контроля качества используемых биологических материалов/объектов становится невоспроизводимость результатов биомедицинских исследований [7].
При этом стоит отметить, что ведущие международные издательства American Association for Cancer Research (AACR) Publications, Society for Endocrinology Journals, Endocrine Society Journals и Nature Publishing Group (NPG), а также такие журналы, как BioTechniques, Cell Biochemistry and Biophysics, In Vitro Cellular & Developmental Biology — Animal, International Journal of Cancer, PLOS ONE, Journal of Molecular Biology и др.2, 3, сформулировали требования по предоставлению информации об аутентификации и чистоте используемых в исследовании клеточных линий [8]. Аналогичные требования/рекомендации при рассмотрении заявок на финансирование предъявляются и ведущими мировыми исследовательскими центрами4.
Существует ряд доступных методов, которые предоставляют информацию о качестве клеточных линий: кариотипирование, профилирование коротких тандемных повторов (short tandem repeat, STR) [9-11], профилирование однонуклеотидных полиморфизмов (SNP) [12][13], использование видоспецифичных праймеров, штрих-кодирование дезоксирибонуклеиновой кислоты (ДНК), высокопроизводительное секвенирование [14-16], СО1 бар-кодирование5. Метод STR-профилирования зарекомендовал себя в качестве надежного и воспроизводимого варианта [17-19] и занял ведущие позиции, в связи с чем Американским национальным институтом стандартов (The American National Standards Institute, ANSI) и Американской коллекцией типовых культур (American Type Culture Collection, ATCC) был разработан стандарт аутентификации клеточных линий человека ASN-0002 (Authentication of Human Cell Lines: Standardization of STR Profiling). Данный документ содержит информацию об актуальности вопроса, значимости аутентификации клеточных линий при проведении биомедицинских исследований, подробный обзор методик, обсуждение их преимуществ и недостатков.
В основе метода лежит анализ коротких тандемных повторов ряда полиморфных STR-локусов, амплифицируемых с использованием коммерчески доступных наборов праймеров, таких как The GlobalFiler (Life Technologies, США), Investigator 24plex QS (Qiagen, Германия), PowerPlex Fusion 6C (Promega, США) или COrDIS "ЭКСПЕРТ 26" (Гордиз, Россия). Набор Investigator 24plex QS содержит ПЦР (полимеразная цепная реакция)-контроль, позволяющий получить дополнительную информацию о качестве ДНК пробы и менее восприимчив к ингибиторам [20]. Набор PowerPlex Fusion 6C также демонстрирует толерантность к ПЦР-ингибиторам, а для The GlobalFiler характерно лучшее восстановление аллелей из образцов ДНК с низким содержанием матрицы [20].
Количество идентифицируемых маркеров варьируется от 24 (GlobalFiler) до 27 (PowerPlex Fusion 6C [21]), однако в составе всех наборов представлены 20 основных локусов CODIS (Combined DNA Index System) и DYS391.
Далее при электрофоретическом разделении с лазер-индуцированной флуоресцентной детекцией продукты ПЦР анализируются одновременно со стандартами размера. Результатом является простой числовой код, соответствующий длинам фрагментов, амплифицированных в каждом локусе. Описанные наборы для STR-профилирования демонстрируют высокие показатели качества полученных результатов, что позволяет взаимно заменять их в лабораторной практике и сравнивать независимо полученные STR-профили. Применяя этот метод к клеточным линиям, лаборатория может, как проверить подлинность коммерческих клеточных линий, так и сформировать базу данных своих линий. При апробации метода в рамках проекта "Создание и развитие биоресурсной коллекции генетически и фенотипически охарактеризованных клеточных линий и первичных опухолей человека", поддержанного грантом Министерства науки и высшего образования РФ, мы проанализировали 37 клеточных линий из Биоресурсной коллекции (БРК) ФГБУ "НМИЦ онкологии им. Н. Н. Блохина". Из них 18 линий анонсированы в международных базах данных и 19 уникальных.
Полученные профили позволили подтвердить возможность использования метода для идентификации образцов коллекции и сформировать базу данных на основе полиморфных STR-локусов уникальных клеточных линий. Кроме того, исследованы возможности метода для обнаружения контаминации клетками других линий.
Материал и методы
Клеточные линии
Получены из БРК клеточных линий и первичных опухолей ФГБУ "НМИЦ онкологии им. Н. Н. Блохина" Минздрава России (таблица 1). Для уникальных клеточных линий, полученных в центре с 01.10.2020г, получено информированное согласие от пациента, составленное по форме, утвержденной Приказом учреждения № 297/П от 01.10.2020г.
Таблица 1
Клеточные линии человека, использованные в работе по определению генетического профиля методом STR-анализа

Примечание: ATCC — American Type Culture Collection (Американская коллекция типовых культур); DSMZ — Deutsche Sammlung von Mikroorganismen und Zellkulturen (немецкая коллекция микроорганизмов и клеточных линий); ECACC — European Collection of Authenticated Cell Cultures (Европейская коллекция аутентифицированных клеточных культур); ИНЦ РАН — Коллекция культур клеток позвоночных Института Цитологии РАН; ИБР РАН — Коллекция клеточных культур Института биологии развития им. Н. К. Кольцова РАН.
Экстракция ДНК
Выделение ДНК из клеток производили с использованием набора Genomic DNA Extraction Kit (Thermo Fisher Scientific, США) в соответствии с протоколом производителя. Количество ДНК оценивали на флуориметре Qubit 4 с использованием набора Qubit™ dsDNA HS Assay Kit (Thermo Fisher Scientific, США).
Приготовление смесей клеточных линий и ДНК С целью определения порога внутривидовой контаминации готовили смеси из предварительно выделенной ДНК клеточных культур mel Pet и mel Kas в 9 вариантах процентного соотношения mel Pet/mel Kas: 96/4; 90/10; 80/20; 70/30; 50/50; 30/70; 20/80; 10/90; 4/96.
После отработки методики на ДНК, была проведена серия экспериментов на смеси культур в тех же соотношениях по количеству клеток для воспроизведения реальных условий клеточной контаминации. Для этого культуры mel Pet и mel Kas смешивали таким образом, чтобы общее количество клеток для выделения ДНК составляло 1 млн. Получение STR — профиля образца Для проведения профилирования на основе коротких тандемных повторов использовали набор COrDIS "ЭКСПЕРТ 26" (Гордиз, Россия) для молекулярно-генетической идентификации личности на основе мультиплексного ПЦР-анализа 26 высоко полиморфных локусов геномной ДНК человека (таблица 2) [22]. Среди них представлены как маркеры стандартных международных панелей локусов: CODIS, EXPANDED CODIS (Expanded Combined DNA Index System), Европейских баз данных на основе ESS (European standard set), включая страны, использующие SE33 как обязательный маркер, так и собственные уникальные маркеры Гордиз.
Таблица 2
Сводная информация о локусах ДНК, присутствующих в составе набора COrDIS "ЭКСПЕРТ 26": для STR-локусов структура единицы повтора приводится в соответствии с рекомендациями Международного Общества Судебных Генетиков (International Society for Forensic Genetics) [22]

Постановку ПЦР проводили в соответствии с рекомендациями производителя, общий объем реакционной смеси составлял 10 мкл. Набор COrDIS "ЭКСПЕРТ 26" позволяет амплифицировать генетический материал в широком диапазоне концентраций ДНК благодаря уникальной технологии нормирования сигнала целевого продукта. В реакционную смесь добавляли 1 мкл геномной ДНК с концентрацией 1-100 нг/мкл. В качестве положительного контроля использовали 6 мкл высокомолекулярной ДНК мужчины в концентрации 0,1 нг/мкл с известным генотипом по всем исследуемым локусам (входит в состав набора). Для приготовления отрицательного контроля использовали деионизированную воду (входит в состав набора).
Разделение продуктов ПЦР осуществляли методом капиллярного электрофореза на генетическом анализаторе с лазер-индуцированной флуоресцентной детекцией (Applied Biosystems 3500xL, США) с использованием капилляров 3500xL Genetic Analyzer 24-Capillary Array (Applied Biosystems, США) длиной 50 см и полимера РОР-7 (Applied Biosystems, США). В качестве внутреннего стандарта для построения кривой подвижности исследуемого образца в каждый капилляр одновременно с образцом вносили стандарт S550 (Гордиз, Россия).
Анализ STR-профиля образца
Анализ флуоресцентных профилей проводили в программе GenMapper 6. Предварительно загрузили панель маркеров и бины, предоставленные производителем набора COrDIS "ЭКСПЕРТ 26". Из-за наличия артефактов анализа рассчитанные программой профили проверялись и корректировались оператором в соответствии с принятыми в международной практике алгоритмами ANSI/ATCC ASN-0002-2011.
Полученные STR-профили для клеточных культур из международных коллекций сравнивали с профилями из баз данных ATCC и DSMZ, на основе % совпадения аллелей маркеров принималось решение о подлинности культуры или о повторном проведении анализа образца.
Процент соответствия рассчитывали по формуле [23]:
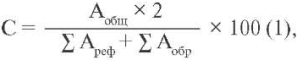
где С — соответствие, %; Аобщ — число общих аллелей, присутствующих и у референса, и у образца; ƩАреф — суммарное число аллелей референса; ƩАобр — суммарное число аллелей образца.
При исследовании возможности метода для обнаружения внутривидовой контаминации использовали клеточные линии mel Pet и mel Kas, для которых идентифицировали 9 общих аллелей, а процент соответствия, рассчитанный по формуле (1), составил 27,7%. Это сказывается на эффективности выявления аллелей контаминирующей линии при построении аналитических диаграмм. Для устранения такого влияния перед построением диаграмм общие для этих линий аллели исключали из анализа.
Результаты и обсуждение
1. Сравнение STR-профилей, полученных для культур, анонсированных в международных базах данных
Профилирование 18 клеточных линий (таблица 1), анонсированных в международных базах данных, показало, что профили 15 линий на 100% совпадали с заявленными в базах. Три культуры: 5637, MCF7 и PANC-1 имели STR-профили, совпадающие на 96,6, 96,3 и 95,8%, соответственно (таблица 3). Для 16 линий провели сравнение профилей с базой ATCC, информация по двум линиям (Efo-21 и PANC-1) в ней отсутствовала, поэтому использовали данные базы DSMZ (Deutsche Sammlung von Mikroorganismen und Zellkulture).
Таблица 3
Сравнение экспериментально полученных генетических профилей клеточных линий 5637, MCF7 и PANC-1 с референсными STR-профилями, аннотированными в базах данных ATCC (A) и DSMZ (Б)

Примечание: STR-профили получены с использованием набора COrDIS "ЭКСПЕРТ 26"; % совпадения, полученного в ходе эксперимента STR-профиля с профилем-референсом из базы данных, рассчитан по формуле (1); жирным шрифтом выделены экспериментально полученные аллели, по которым наблюдается неполное совпадение с референсными профилями из баз данных.
Согласно рекомендациям ANSI/ATCC ASN0002-2011 для подтверждения подлинности клеточной линии соответствие должно составлять 80100%. Совпадение на 80-50% требует повторного анализа культуры, а значение параметра ≤50% подтверждает отличное от референса происхождение клеток. Таким образом, в рамках настоящей работы подтверждена подлинность 18 клеточных линий, анонсированных в международных базах.
2. Формирование внутренней базы данных STR-профилей для уникальных культур, полученных в "НМИЦ онкологии им. Н. Н. Блохина"
Для формирования базы данных генетических профилей уникальных образцов исследовали 19 клеточных линий (таблица 1). Для каждого образца повторяли исследование не <3 раз и только при полной воспроизводимости результатов клеточной линии присваивали профиль. Полученные профили позволили заложить начало базы данных БРК центра, информация вносится в паспорт клеточной линии, и предоставляется пользователям по запросу.
При проверке новой клеточной линии на контаминацию клетками других линий полученный профиль сравнивали с референсом. В качестве референсного использовали STR-профиль ДНК, выделенной из фрагмента ткани пациента, использованного в последующем для получения новой клеточной линии. Данный профиль можно использовать только для выявления контаминации за счет обнаружения новых аллелей, но не в качестве основного, поскольку в процессе возможны различные хромосомные перестройки, приводящие к изменению STR-профиля, например, потеря гетерозиготности, как это происходит в случае линии mel Pet.
3. Определение порогов контаминации другими клеточными линиями
Как уже обсуждалось выше, контаминация другой клеточной линией является широко распространенной проблемой. Стратегия контроля качества образцов, принятая в БРК клеточных линий и первичных опухолей "НМИЦ онкологии им. Н. Н. Блохина", подразумевает строгий контроль контаминации, в т.ч. внутривидовой. Метод STR-профилирования прекрасно подходит для этих целей, т.к. каждая из клеточных линий имеет свой уникальный генетический профиль, а в смеси клеточных линий окончательный результат будет отражать комбинацию профилей присутствующих клеток. Нашей задачей было определить параметры оценки контаминации и установить их пределы обнаружения. Хотя интерпретация пиков в одном локусе может быть сложной и неоднозначной, анализ 26 различных локусов идентифицирует пики, которые четко указывают на наличие загрязняющей клеточной линии.
Обнаружение контаминации основано на выявлении в STR-профиле нехарактерных для данной клеточной культуры дополнительных аллелей6. Результаты, полученные при изучении смесей с разным соотношением ДНК двух клеточных линий mel Pet и mel Kas, представлены на рисунке 1.

Рис. 1 Диаграмма распределения аллелей в STR-профилях смесей ДНК с различным соотношением клеточных культур mel Pet и mel Kas.
Примечание: на рисунке представлена диаграмма, иллюстрирующая распределение уникальных аллелей в STR-профилях, полученных для смесей с различным %-ным содержанием ДНК клеточных культур mel Pet и mel Kas. Синим цветом обозначен процент содержания аллелей, уникальных для культуры mel Pet, зеленым — уникальных для культуры mel Kas. Оранжевой линией соединены рассчитанные значения %-го обнаружения уникальных аллелей контаминанта в профиле преобладающей клеточной культуры.
В смесях ДНК уже при 4%-ной контаминации mel Pet в STR-профиле проявляются дополнительные аллели для 4-х маркеров, при 10%-ной контаминации детектируется более половины аллелей посторонней культуры, а при 20%-ном содержании удается практически на 100% восстановить профиль и идентифицировать культуру-контаминант. При 20%-ом содержании mel Kas проявляется только половина специфичных аллелей.
В эксперименте по смешиванию двух клеточных культур получены схожие результаты (рисунок 2). Из рисунка 2 видно, что уже при 4%-ной контаминации на STR-профиле культуры наблюдаются посторонние дополнительные аллели, однако установление полного профиля культуры-контаминанта достигается при 30%-ной контаминации.

Рис. 2 Диаграмма распределения аллелей в STR-профилях смесей клеточных культур mel Pet и mel Kas с различным соотношением клеток.
Примечание: на рисунке представлена диаграмма, иллюстрирующая распределение уникальных аллелей в STR-профилях, полученных для смесей с различным %-ным содержанием клеток культур mel Pet и mel Kas. Синим цветом обозначен процент содержания аллелей, уникальных для культуры mel Pet, зеленым — уникальных для культуры mel Kas. Оранжевой линией соединены рассчитанные значения %-го обнаружения уникальных аллелей контаминанта в профиле преобладающей клеточной культуры.
Культура mel Pet проявляет тенденцию к более раннему обнаружению в смеси. Этот эффект ярко выражен в ДНК смесях, но также сохраняется в смесях клеточных культур. Возможной причиной такого явления могут быть особенности клеточной линии, связанные с плоидностью или с выраженной аллельной гомозиготностью культуры, хорошо заметной в STR-профиле. В таком случае при появлении гетерозиготности или третьего аллеля по отдельным маркерам легче заподозрить контаминацию.
В ходе анализа результатов эксперимента выяснилось, что настройки программного обеспечения (ПО) не позволяют по числовому коду, определяющему генетический профиль, обнаружить <20-30% контаминанта, в то время как дополнительная обработка результатов эксперимента, основанная на анализе электрофореграмм, позволяет оператору детектировать отдельные аллели уже при 4%-ом содержании контаминирующей линии. На рисунке 3 представлены примеры изменения высоты пиков при разных соотношениях клеточных линий для 6 маркеров: D22S1045, D18S51, D16S539, D8S1179, CSF1PO и D6S1043. Пик аллеля маркера D18S51, характерный для клеточной линии mel Kas, появляется при содержании всего 4% линии-контаминанта, однако ПО расценивает его как шум или артефакт. Высота данного пика относительно основного, с одной стороны, невелика (1121 RFU к 13862 RFU), но при этом превышает пороговые значения сигнала (>500 RFU) — двусмысленная ситуация не позволяет оператору точно идентифицировать дополнительный аллель. В этом случае необходимо обратить внимание на электрофореграммы всех маркеров и, при наличии аналогичной картины хотя бы по некоторым из них, принять решение о статусе рассматриваемого пика. При 10%-ой контаминации линией mel Kas пик по данному маркеру дает оператору возможность с большей уверенностью предположить появление дополнительного аллеля и проверить гипотезу анализом пиков по другим маркерам. ПО определяет аллель по данному маркеру при 20%-ом содержании контаминирующей линии, а по маркерам D16S539 и D8S1179 — только при 50%-ом содержании линии mel Kas.

Рис. 3 Электрофореграммы смесей культур клеток mel Pet и mel Kas.
Примечание: на рисунке представлены профили клеток mel Pet и mel Kas и их смесей по 6 маркерам, %-ное содержание клеток линии mel Pet показано синим цветом, mel Kas — красным. Кружками отмечены пики, которые программа не считывает за сигнал, а оператор может определить как проявление дополнительного аллеля и контаминации. Числовые значения выдает ПО: синим цветом — аллели, характерные для линии mel Pet, красным — для линии mel Kas, черным — общие для двух линий.
Другой интересный пример представлен на рисунке 3 для маркера CSF1PO. Для линий mel Pet и mel Kas аллели по этому маркеру находятся очень близко, поэтому появление дополнительных пиков, характерных для контаминирующих клеток, при 30%-ой контаминации может быть расценено оператором либо как широко распространенный артефакт (статтеры), либо в качестве самостоятельных аллелей, в то время как ПО определяет их как дополнительные аллели при 20-30% контаминации.
Эти примеры свидетельствуют, что, с одной стороны, данный метод более чувствителен по сравнению с очевидными признаками внутривидовой контаминации: изменение морфологии и скорости роста, изменение результатов воздействия различными препаратами и т.д. С другой стороны, требуется сформулировать общие рекомендации по настройке фильтра аллелей в ПО и разработать четкий алгоритм действий оператора при анализе результатов STR-профилирования.
Заключение
Идентификация клеточных линий необходима для обеспечения надлежащего качества исследований и воспроизводимости результатов. Использование метода профилирования на основе коротких тандемных повторов является "золотым стандартом" для подтверждения подлинности клеточных линий человека, хранящихся в биобанках.
Полученные результаты показали соответствие профилей коммерческих клеточных линий с данными в международных базах, что подтверждает возможность использования набора COrDIS "ЭКСПЕРТ 26" (Гордиз, Россия) для идентификации образцов коллекции.
В рамках настоящей работы были получены профили уникальных линий и заложено начало собственной генетической базы БРК, что обеспечивает контроль качества образца на всех этапах существования клеточной линии в коллекции и гарантирует пользователям предоставление материала надлежащего качества.
Исследования на выявление предела обнаружения контаминации другой линией показали, что при содержании в общем пуле даже 4% культуры-контаминанта удается выявить ее отдельные аллели. Данные результаты свидетельствуют о более высокой чувствительности метода по сравнению с очевидными признаками внутривидовой контаминации: изменение морфологии и скорости роста, изменение результатов воздействия различными препаратами и т.д.
Отношения и деятельность: Работа выполнена при финансовой поддержке Министерства науки и высшего образования Российской Федерации в рамках научно-исследовательской работы № 07515-2021-1060 «Создание и развитие биоресурсной коллекции генетически и фенотипически охарактеризованных клеточных линий и первичных опухолей человека».
1. ICLAC Register of Misidentified Cell Lines. https://iclac.org/databases/cross-contaminations/.
2. List of Journals requiring Cell Line Authentication. https://worldwide.promega.com/resources/guides/cell-biology/cell-lineauthentication/.
3. ATCC Cell Line Authentication Publication Requirements. https://www.atcc.org/the-science/authentication/cell-line-authenticationpublicationrequirements.
4. Enhancing Reproducibility through Rigor and Transparency. https://grants.nih.gov/grants/guide/notice-files/NOT-OD-15-103.html.
5. ATCC SDO Species-Level Identification of Animal Cells through Mitochondrial Cytochrome c Oxidase Subunit 1 (CO1) DNA Barcodes. ATCC SDO document ASN-0003. Manassas, VA: ATCC Standards Development Organization, 2015.
6. Authenticating human cell lines using CLA IdentiFiler and CLA GlobalFiler kits on capillary electrophoresis platforms. Thermo Fisher Scientific Application Note. 2020. https://assets.thermofisher.com/TFS-Assets/GSD/Application-Notes/authenticating-human-celllinesstr-kits-capillary-electrophoresis-application-note.pdf. (9 августа 2022).
Список литературы
1. Allen M, Bjerke M, Edlund H, et al. Origin of the U87MG glioma cell line: Good news and bad news. Sci Transl Med. 2016;8(354):354re3. doi:10.1126/scitranslmed.aaf6853.
2. Elgui de Oliveira D, Marques CS, Losi VC. "Cell identity" crisis: Another call for immediate action. Cancer Lett. 2016;381(1):122- 3. doi:10.1016/j.canlet.2016.07.031.
3. Horbach SPJM, Halffman W. The ghosts of HeLa: How cell line misidentification contaminates the scientific literature. PLoS One. 2017;12(10):e0186281. doi:10.1371/journal.pone.0186281.
4. Capes-Davis A, Theodosopoulos G, Atkin I, et al. Check your cultures! A list of cross-contaminated or misidentified cell lines. Int J Cancer. 2010;127(1):1-8. doi:10.1002/ijc.25242.
5. Drexler HG, Dirks WG, MacLeod RA, et al. False and mycoplasma-contaminated leukemia-lymphoma cell lines: time for a reappraisal. Int J Cancer. 2017;140(5):1209-14. doi:10.1002/ijc.30530.
6. Neimark J. Line of attack. Science. 2015;347(6225):938-40. doi:10.1126/science.347.6225.938.
7. Jarvis MF, Williams M. Irreproducibility in Preclinical Biomedical Research: Perceptions, Uncertainties, and Knowledge Gaps. Trends Pharmacol Sci. 2016;37(4):290-302. doi:10.1016/j.tips.2015.12.001.
8. Geraghty RJ, Capes-Davis A, Davis JM, et al. Guidelines for the use of cell lines in biomedical research. Br J Cancer. 2014; 111(6):1021-46. doi:10.1038/bjc.2014.166.
9. Visconti P, Parodi F, Parodi B, et al. Short tandem repeat profiling for the authentication of cancer stem-like cells. Int J Cancer. 2021;148(6):1489-98. doi:10.1002/ijc.33370.
10. Almeida JL, Dakic A, Kindig K, et al. Interlaboratory study to validate a STR profiling method for intraspecies identification of mouse cell lines. PLoS One. 2019;14(6):e0218412. doi:10.1371/journal.pone.0218412.
11. Gu M, Yang M, He J, et al. A silver lining in cell line authentication: Short tandem repeat analysis of 1373 cases in China from 2010 to 2019. Int J Cancer. 2022;150(3):502-8. doi:10.1002/ijc.33789.
12. Castro F, Dirks WG, Fähnrich S, et al. High-throughput SNPbased authentication of human cell lines. Int J Cancer. 2013;132(2):308-14. doi:10.1002/ijc.27675.
13. Didion JP, Buus RJ, Naghashfar Z, et al. SNP array profiling of mouse cell lines identifies their strains of origin and reveals crosscontamination and widespread aneuploidy. BMC Genomics. 2014;15(1):847. doi:10.1186/1471-2164-15-847.
14. Fasterius E, Raso C, Kennedy S, et al. A novel RNA sequencing data analysis method for cell line authentication. PLoS One. 2017;12(2):e0171435. doi:10.1371/journal.pone.0171435.
15. Chen X, Qian W, Song Z, et al. Authentication, characterization and contamination detection of cell lines, xenografts and organoids by barcode deep NGS sequencing. NAR Genom Bioinform. 2020;2(3):lqaa060. doi:10.1093/nargab/lqaa060.
16. Lung O, Candlish R, Nebroski M, et al. High-throughput sequencing for species authentication and contamination detection of 63 cell lines. Sci Rep. 2021;11(1):21657. doi:10.1038/s41598-021-00779-5.
17. Masters JR, Thomson JA, Daly-Burns B, et al. Short tandem repeat profiling provides an international reference standard for human cell lines. Proc Natl Acad Sci USA. 2001;98(14):8012-7. doi:10.1073/pnas.121616198.
18. Мельникова Е. В., Меркулова О.В., Меркулов В.А. и др. Идентификация клеточных линий человека с использованием метода генотипирования короткими тандемными повторами: мировая практика. Биофармацевтический журнал. 2015;7(6):3-10. EDN VRRBXF.
19. Хорольский М.Д., Семенова И.С., Мельникова Е.В. и др. Применение метода коротких тандемных повторов для аутентификации клеточных линий. БИОпрепараты. Профилактика, диагностика, лечение. 2019;19(4):251-60. doi:10.30895/2221-996X-2019-19-4-251-260.
20. Tan JYY, Tan YP, Ng S, et al. A preliminary evaluation study of new generation multiplex STR kits comprising of the CODIS core loci and the European Standard Set loci. J Forensic Leg Med. 2017;52:16-23. doi:10.1016/j.jflm.2017.07.017.
21. Cho WC, Jung JK, Cho Y, et al. Validation and assessment of the Investigator® 24plex QS kit for forensic casework application: Comparison with the PowerPlex® fusion system and GlobalFiler™ PCR amplification kits. Leg Med (Tokyo). 2021;52:101902. doi:10.1016/j.legalmed.2021.101902.
22. Bär W, Brinkmann B, Budowle B, et al. DNA recommendations. Further report of the DNA Commission of the ISFH regarding the use of short tandem repeat systems. International Society for Forensic Haemogenetics. Int J Legal Med. 1997;110(4):175-6. doi:10.1007/s004140050061.
23. Tanabe H, Takada Y, Minegishi D, et al. Сell line individualization by STR multiplex system in the cell bank found crosscontamination between ECV304 and EJ-1/T24. Tissue culture research communications: the journal of experimental & applied cell culture research. 1999;18:329-38. doi:10.11418/jtca1981.18.4_329.
Об авторах
Е. Н. КособоковаРоссия
Кандидат биологических наук, ведущий научный сотрудник лаборатории трансгенных препаратов НИИ экспериментальной диагностики и терапии опухолей
Москва
А. А. Мальченкова
Россия
Младший научный сотрудник лаборатории трансгенных препаратов НИИ экспериментальной диагностики и терапии опухолей
Москва
Н. А. Калинина
Россия
Младший научный сотрудник лаборатории трансгенных препаратов НИИ экспериментальной диагностики и терапии опухолей
Москва
В. С. Косоруков
Россия
Кандидат биологических наук, заведующая лабораторией трансгенных препаратов, директор НИИ экспериментальной диагностики и терапии опухолей
Москва
Дополнительные файлы
Рецензия
Для цитирования:
Кособокова Е.Н., Мальченкова А.А., Калинина Н.А., Косоруков В.С. Использование метода профилирования на основе коротких тандемных повторов для подтверждения подлинности клеточных линий в биобанках. Кардиоваскулярная терапия и профилактика. 2022;21(11):3386. https://doi.org/10.15829/1728-8800-2022-3386
For citation:
Kosobokova E.N., Malchenkova A.A., Kalinina N.A., Kosorukov V.S. Using short tandem repeat profiling to validate cell lines in biobanks. Cardiovascular Therapy and Prevention. 2022;21(11):3386. (In Russ.) https://doi.org/10.15829/1728-8800-2022-3386
JATS XML

























































