Перейти к:
Использование шкал генетического риска для дифференциальной диагностики у лиц с клиническим диагнозом семейная гиперхолестеринемия
https://doi.org/10.15829/1728-8800-2024-4251
EDN: UYQMBM
Аннотация
Цель. Оценить полигенный вклад в уровни холестерина (ХС) липопротеинов низкой плотности (ЛНП) и общего ХС у пациентов с клиническим диагнозом гетерозиготная семейная гиперхолестеринемия (СГХС) с помощью шкал генетического риска (ШГР); исследовать возможность предсказания направительного диагноза СГХС среди неносителей патогенных вариантов на основе данных ШГР.
Материал и методы. В исследовании были использованы результаты генетического тестирования популяционной выборки из Ивановской области (n=1673) и пациентов ФГБУ "НМИЦ ТПМ" Минздрава России с диагнозом СГХС (n=353). Исследование включало три различных ШГР для ХС ЛНП и три ШГР для общего ХС.
Результаты. В рамках исследования оценена возможность использования ШГР для российской популяции: процент объясненной дисперсии для ШГР ХС ЛНП составил от 4,54 до 6,23%, для общего ХС — от 2,74 до 5,98%. Было показано, что значения ШГР значимо (p<0,001) различаются между тремя группами: популяционной выборкой из Ивановской области, пациентами с клиническим диагнозом СГХС, являющимися носителями и неносителями известных патогенных вариантов в генах LDLR, APOB и PCSK9. Значения ШГР также можно использовать для дифференциальной диагностики у лиц с клиническим диагнозом СГХС и для выявления лиц с полигенной гиперхолестеринемией.
Заключение. Впервые были исследованы предсказательные возможности ШГР для ХС ЛНП и общего ХС среди российских пациентов с СГХС.
Ключевые слова
Для цитирования:
Зайченока М., Мешков А.Н., Киселева А.В., Ершова А.И., Сотникова Е.А., Жарикова А.А., Вяткин Ю.В., Михайлина В.И., Букаева А.А., Покровская М.С., Раменский В.Е., Драпкина О.М. Использование шкал генетического риска для дифференциальной диагностики у лиц с клиническим диагнозом семейная гиперхолестеринемия. Кардиоваскулярная терапия и профилактика. 2024;23(12):4251. https://doi.org/10.15829/1728-8800-2024-4251. EDN: UYQMBM
For citation:
Zaychenoka M., Meshkov A.N., Kiseleva A.V., Ershova A.I., Sotnikova E.A., Zharikova A.A., Vyatkin Yu.V., Mikhailina V.I., Bukaeva A.A., Pokrovskaya M.S., Ramenskiy V.E., Drapkina O.M. Use of polygenic risk scores for differential diagnostics for patients with clinical diagnosis of familial hypercholesterolemia. Cardiovascular Therapy and Prevention. 2024;23(12):4251. (In Russ.) https://doi.org/10.15829/1728-8800-2024-4251. EDN: UYQMBM
Введение
Семейная гиперхолестеринемия (СГХС) является наследственным заболеванием, сопровождающимся повышением уровня холестерина (ХС) липопротеинов низкой плотности (ЛНП) и ранним развитием атеросклероза и его осложнений [1].
Различают гетерозиготную и гомозиготную формы заболевания, вызываемого преимущественно вариантами нуклеотидной последовательности (ВНП) в генах LDLR, APOB и PCSK9 [2]. Распространенность СГХС в разных регионах Российской Федерации составляет от 1/108 до 1/173 человека [3]. Среди пациентов с клинически диагностированной гетерозиготной СГХС удается идентифицировать ВНП, являющиеся причиной заболевания в 40-70% случаев [4]. В остальных случаях заболевание имеет полигенную природу [5]. Показано, что прогноз у лиц с клиническим диагнозом гетерозиготная СГХС и выявленными причинными вариантами в генах LDLR, APOB и PCSK9 хуже, чем у лиц с полигенным механизмом развития гиперхолестеринемии (ГХС) [6].
Оценить вклад частых ВНП в развитие фенотипа сложно, т.к. эти ВНП по отдельности вносят лишь небольшой вклад в итоговый фенотип [7]. Полногеномный поиск ассоциаций (genome-wide association study, GWAS) позволяет определить вклад индивидуальных ВНП и на основе результатов GWAS разрабатывать шкалы генетического риска (ШГР) [8], которые позволяют количественно оценить полигенный вклад в развитие фенотипа [9].
В литературе описано множество различных ШГР, для которых показана связь с ГХС. Trinder M, et al. (2020) [10] в своем исследовании разработали ШГР из 28 ВНП и показали для нее значимую ассоциацию с уровнем ХС ЛНП и риском раннего развития атеросклероза в когорте из пациентов с ГХС. Khera AV, et al. (2018) [11] показали, что индивидуумы, попадающие в 8% самых высоких значений ШГР для ишемической болезни сердца (ИБС) из >6000000 ВНП, подвержены риску ее развития, схожему с риском при наличии моногенного ВНП, связанного с СГХС. Wu H, et al. (2021) [12] в своей работе представили ШГР из >8000 ВНП для ХС ЛНП для больных СГХС и показали, что у носителей известных патогенных ВНП в генах, связанных с СГХС, значения ШГР значимо ниже, чем у неносителей.
Целью настоящей работы была оценка полигенного вклада в уровни ХС ЛНП и общего ХС у пациентов с клиническим диагнозом гетерозиготная СГХС с помощью шкал генетического риска (ШГР), а также исследование возможности предсказания направительного диагноза СГХС среди неносителей патогенных вариантов на основе данных ШГР.
Материал и методы
Выборка. В исследовании использовались две выборки:
- Выборка из участников эпидемиологического исследования ЭССЕ-РФ (Эпидемиология сердечно-сосудистых заболеваний в регионах России) из Ивановской области (ЭССЕ-Иваново), состоящая из 1673 человек в возрасте 24-66 лет [13].
- Выборка больных СГХС, состоящая из 353 человек в возрасте 18-78 лет, наблюдаемых в рамках регистра РЕНЕССАНС (Регистр пациентов с семейной гиперхолестеринемией и пациентов очень высокого сердечно-сосудистого риска с недостаточной эффективностью проводимой гиполипидемической терапии) в ФГБУ "НМИЦ ТПМ" Минздрава России [14]. Диагноз был поставлен согласно критериям Dutch Lipid Clinic Network, рекомендациям Европейского общества по атеросклерозу [14].
Сбор и хранение биообразцов выполняли согласно регламенту биобанкирования в Биобанке ФГБУ "НМИЦ ТПМ" Минздрава России (г. Москва) [15][16].
Исследование одобрено Независимым Этическим Комитетом ФГБУ "НМИЦ ТПМ" Минздрава России (номера протоколов 07-03/12 от 03.07.2012 и 04-04/17 от 06.06.2017). Все участники дали письменное информированное согласие.
Генетический анализ. Выделение дезоксирибонуклеиновой кислоты (ДНК) из цельной крови проводилось с использованием набора QIAamp DNA Blood Mini Kit (Qiagen, Германия). Для оценки качества и определения концентрации выделенной ДНК использовали флуориметр Qubit 4.0 (Thermo Fisher Scientific, США). Подготовка библиотек для секвенирования ДНК была проведена с помощью набора SeqCap EZ Prime Choice Library (Roche, Швейцария).
Для каждого участника исследования было проведено секвенирование следующего поколения. Для секвенирования ДНК участников выборки ЭССЕ-Иваново была использована таргетная панель, включающая 242 гена и 2042 ВНП [13]. Секвенирование было проведено на приборе NextSeq550 (Illumina, США) с получением парноконцевых прочтений длиной 75 или 150 п.н. Все этапы секвенирования были проведены в соответствии с протоколами производителей.
Биоинформатический и статистический анализ. Парноконцевые прочтения в формате fastq были выровнены на референсный геном GRCh38. Обработка данных и оценка контроля качества выполнялись с помощью специально разработанного пайплайна [13] основанного на GATK 3.8 [17]. Аннотацию ВНП и коротких инсерций и делеций осуществляли с помощью ENSEMBL Variant Effect Predictor (v. 100) [18] и базы данных ClinVar [19]. Из выборок были удалены родственные образцы на основе анализа пакетом PLINK v. 1.90 [20]. Из пары родственников оставляли старшего по возрасту.
Для расчета ШГР использовались данные GWAS по ХС ЛНП и общему ХС из публикаций Willer CJ, et al. (2013) [21] и Selvaraj MS, et al. (2022) [22] и данные ШГР Xu Y, et al. (2023) [23]. Шкалы Willer CJ, et al. (2013) [21] широко используются в литературе для изучения сердечно-сосудистых заболеваний. Шкалы Selvaraj MS, et al. (2022) [22] разработаны на основе самого большого GWAS на основе данных полногеномного секвенирования — использовано >66000 образцов. Шкалы Xu Y, et al. (2023) [23] взяты из первого атласа ШГР для мульти-омиксных данных. Для расчета ШГР использовались варианты из оригинального исследования, идентифицированные в выборках. В случае Willer CJ, et al. (2013) [21] были идентифицированы все варианты, для шкал Selvaraj MS, et al. (2022) [22] и Xu Y, et al. (2023) [23] было идентифицировано от 9 до 18%. ШГР рассчитывали с помощью собственного скрипта, который на основе данных о рисковом аллеле и оценке эффекта рискового аллеля производил расчет значения шкалы как суммы произведений оценок эффекта и количества рисковых аллелей. Статистический анализ проводили, используя среду R 4.1.2 с открытым исходным кодом. Для дальнейшей работы значения ШГР приводили к среднему 0 и стандартному отклонению 1, используя Z-стандартизацию. Для оценки качества ШГР использовали коэффициент детерминации (R²), полученный из линейной регрессии. Оценка предсказательной возможности бинарных признаков была проведена на основе значений площади под ROC-кривой (AUC), полученных из логистической регрессии. Различия между двумя независимыми выборками для непрерывных параметров оценивали, используя тест Манна-Уитни-Вилкоксона; между тремя независимыми выборками — тест Краскела-Уоллиса, для категориальных признаков — критерий χ². Уровень статистической значимости равнялся 0,05.
Результаты
Клиническая характеристика участников представлена в таблице 1. Выборка пациентов с СГХС содержала меньшую долю мужчин и была значимо старше выборки ЭССЕ-Иваново. Очевидно, что уровни ХС ЛНП и общего ХС значимо различались между выборками.
Таблица 1
Клиническая характеристика участников исследования
|
Показатель |
ЭССЕ-Иваново (n=1673) |
СГХС (n=353) |
р |
|
Мужской пол, n (%) |
624 (37,3) |
99 (28,0) |
<0,001 |
|
Возраст, лет, Ме [ Q25; Q75] |
50 [ 40; 57] |
58 [ 50; 63] |
<0,001 |
|
ХС ЛНП, ммоль/л, Ме [ Q25; Q75] |
3,50 [ 2,73; 4,30] |
5,52 [ 4,63; 6,52] |
<0,001 |
|
Общий ХС, ммоль/л, Ме [ Q25; Q75] |
5,65 [ 4,87; 6,45] |
7,9 [ 6,75; 8,87] |
<0,001 |
Примечание: ЛНП — липопротеины низкой плотности, СГХС — семейная гиперхолестеринемия, ХС — холестерин, ЭССЕ-РФ — Эпидемиология сердечно-сосудистых заболеваний в регионах России (из Ивановской области — ЭССЕ-Иваново).
Перед дальнейшим анализом была проведена оценка применимости данных ШГР к выборке ЭССЕ-Иваново. Для этого оценивали силу ассоциации значений ШГР с соответствующими фенотипами (ХС ЛНП, общий ХС) и рассчитывали значения R² (таблица 2). Полученные значения R² варьировались от 2,74% для общего ХС [23] до 6,23% для ХС ЛНП [22]. Для всех ШГР в таблице 2 показаны значимые ассоциации (р<0,05).
Таблица 2
Оценка применимости ШГР к российской популяции
|
ШГР |
Фенотип |
Количество ВНП |
R², % |
95% ДИ R² |
р |
|
Willer CJ, et al. (2013) [ 21] |
ХС ЛНП |
57 |
4,54 |
[ 2,81; 6,68] |
6,85×10⁻²¹ |
|
Общий ХС |
73 |
5,98 |
[ 4,01; 8,31] |
3,40×10⁻²⁸ |
|
|
Selvaraj MS, et al. (2022) [ 22] |
ХС ЛНП |
48 |
6,23 |
[ 4,31; 8,56] |
2,37×10⁻²⁸ |
|
Общий ХС |
51 |
4,45 |
[ 2,81; 6,36] |
3,27×10⁻²¹ |
|
|
Xu Y, et al. (2023) [ 23] |
ХС ЛНП |
35 |
5,09 |
[ 3,34; 7,21] |
2,68×10⁻²³ |
|
Общий ХС |
34 |
2,74 |
[ 1,46; 4,41] |
1,53×10⁻¹³ |
Примечание: ВНП — варианты нуклеотидной последовательности, ДИ — доверительный интервал, ЛНП — липопротеины низкой плотности, ХС — холестерин, ШГР — шкала генетического риска, R2 — коэффициент детерминации.
В дальнейшем анализе в качестве ковариат во всех моделях использовали пол и возраст. Выборки ЭССЕ-Иваново и выборка пациентов с СГХС были объединены для дальнейшего анализа, первая использовалась в качестве контрольной. В выборке пациентов с СГХС были определены носители (n=116, 32,8%) и неносители (n=237, 67,2%) патогенных ВНП в генах APOB, LDLR и PCSK9. Носителей патогенных ВНП в указанных генах в выборке ЭССЕ-Иваново выявлено не было. Сравнение пациентов с патогенным ВНП и без него по клиническим характеристикам показало, что у пациентов без патогенного ВНП обнаружены значимо более низкие уровни ХС ЛНП и общего ХС, реже наблюдались липоидная дуга и ксантомы сухожилий, у меньшей доли пациентов случался инфаркт миокарда, а также они набирали меньше баллов согласно критериям Dutch Lipid Clinic Network. Более того, пациенты без патогенного ВНП были значимо старше. При этом доля мужчин и частота выявления ИБС в обеих выборках значимо не различались (таблица 3). Было проведено сравнение распределений значений ШГР в популяции, среди пациентов с СГХС с патогенным ВНП, и среди пациентов с СГХС без патогенного ВНП тестом Краскела-Уоллиса. Оказалось, что для всех ШГР значения значимо различались хотя бы для двух групп (рисунок 1). В среднем наименьшие значения были достигнуты в выборке ЭССЕ-Иваново, затем в выборке пациентов с клиническим диагнозом СГХС — носителями патогенных ВНП. Наивысшие значения в большинстве случаев достигались в выборке пациентов с клиническим диагнозом СГХС, которые не являлись носителями патогенных ВНП в генах APOB, LDLR и PCSK9.
Таблица 3
Сравнение клинических характеристик между пациентами с патогенным ВНП и без него
|
Показатель |
Пациенты с патогенным ВНП |
Пациенты без патогенного ВНП |
р |
|
Мужчин, n (%) |
40 (34,5) |
59 (24,9) |
0,07 |
|
Возраст, лет, Ме [ Q25; Q75] |
52 [ 41; 62] |
58 [ 53; 64] |
<0,001 |
|
ХС ЛНП, ммоль/л, Ме [ Q25; Q75] |
6,24 [ 4,76; 7,65] |
5,45 [ 4,58; 6,17] |
<0,001 |
|
Общий ХС, ммоль/л, Ме [ Q25; Q75] |
8,08 [ 6,72; 9,90] |
7,79 [ 6,79; 8,54] |
0,007 |
|
Ишемическая болезнь сердца, % |
28,2 |
36,7 |
0,149 |
|
Инфаркт, % |
19,8 |
8,6 |
0,012 |
|
Липоидная дуга, % |
25,7 |
14,2 |
0,018 |
|
Ксантомы сухожилий, % |
55,0 |
20,2 |
<0,001 |
|
Баллы согласно критериям Dutch Lipid Clinic Network, Ме [ Q25; Q75] |
11 [ 6; 15] |
6 [ 3; 8] |
<0,001 |
Примечание: ВНП — варианты нуклеотидной последовательности, ЛНП — липопротеины низкой плотности, ХС — холестерин.
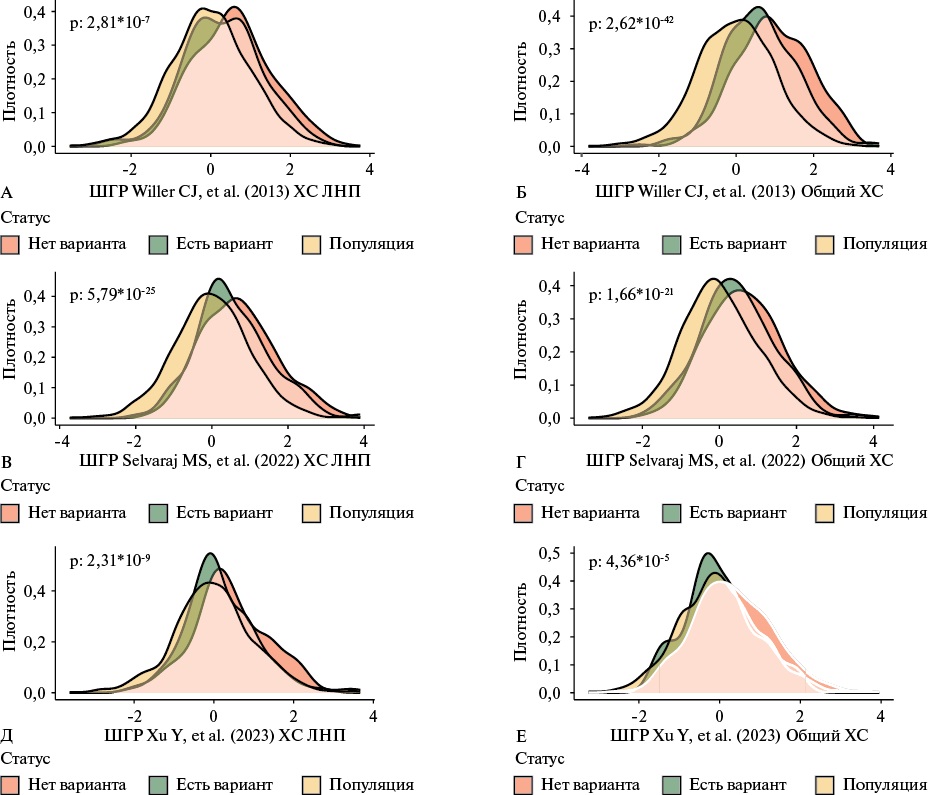
Рис. 1 Сравнение распределений значений ШГР между контрольной группой (ЭССЕ-Иваново) и пациентами с СГХС (с патогенным ВНП и без него). Для каждой ШГР приведены р-значения теста Краскела-Уоллиса. А) ШГР Willer CJ, et al. (2013) [21] для ХС ЛНП; Б) ШГР Willer CJ, et al. (2013) [21] для общего ХС; В) ШГР Selvaraj MS, et al. (2022) [ 22] для ХС ЛНП; Г) ШГР Selvaraj MS, et al. (2022) [22] для общего ХС; Д) ШГР Xu Y, et al. (2023) [23] для ХС ЛНП; Е) ШГР Xu Y, et al. (2023) [23] для общего ХС.
Примечание: ВНП — варианты нуклеотидной последовательности, ЛНП — липопротеины низкой плотности, СГХС — семейная гиперхолестеринемия, ХС — холестерин, ШГР — шкала генетического риска, ЭССЕ-РФ — Эпидемиология сердечно-сосудистых заболеваний в регионах России (из Ивановской области — ЭССЕ-Иваново). Цветное изображение доступно в электронной версии журнала.
Для распределений значений ШГР между выборками пациентов с СГХС с патогенным ВНП и без него было проведено сравнение тестом Манна-Уитни-Вилкоксона. Для всех ШГР, кроме предложенной Selvaraj MS, et al. (2022) [22], было показано значимое различие (p<0,05) между значениями ШГР в двух группах (рисунок 2). Рисунок 2 получен из рисунка 1 путем удаления распределений значений ШГР для Иваново для улучшения наглядности.
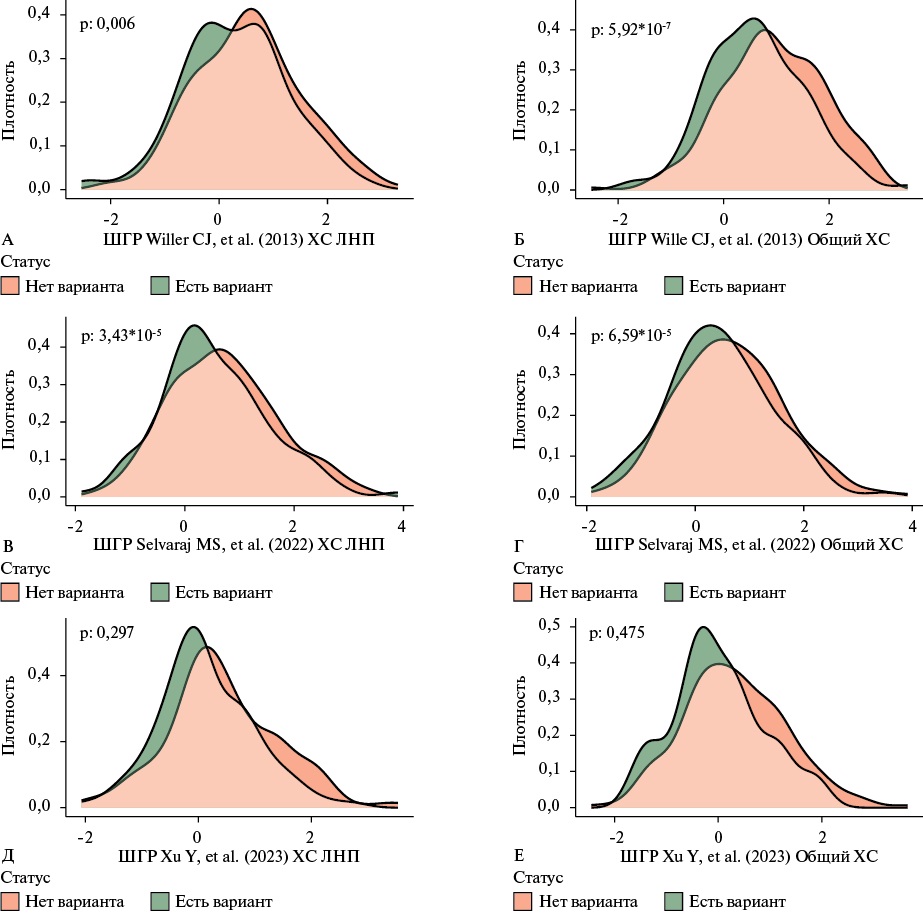
Рис. 2 Сравнение распределений значений ШГР между двумя группами пациентов с СГХС — носителями патогенных ВНП и без них. Для каждой ШГР приведены р-значения теста Манна-Уитни-Вилкоксона. А) ШГР Willer CJ, et al. (2013) [21] для ХС ЛНП; Б) ШГР Willer CJ, et al. (2013) [21] для общего ХС; В) ШГР Selvaraj MS, et al. (2022) [22] для ХС ЛНП; Г) ШГР Selvaraj MS, et al. (2022) [22] для общего ХС; Д) ШГР Xu Y, et al. (2023) [23] для ХС ЛНП; Е) ШГР Xu Y, et al. (2023) [23] для общего ХС.
Примечание: ВНП — варианты нуклеотидной последовательности, ЛНП — липопротеины низкой плотности, СГХС — семейная гиперхолестеринемия, ХС — холестерин, ШГР — шкала генетического риска. Цветное изображение доступно в электронной версии журнала.
На основании полученных результатов можно предположить, что для неносителей известных патогенных вариантов в генах APOB, LDLR и PCSK9 наличие клинического диагноза СГХС можно предсказывать с помощью модели на основе значений ШГР. Для этого были использованы выборки ЭССЕ-Иваново (n=1673) и выборка пациентов с клиническим диагнозом СГХС, не являющихся носителями патогенных ВНП (n=237). В качестве ковариаты использовали пол участников исследования. AUC нулевой модели (без учета ШГР) составил 0,56. Все шкалы позволили создать модели, позволяющие значимо лучше предсказать наличие клинического диагноза СГХС среди неносителей патогенного варианта, чем модель, использующая только данные пола (таблица 4, рисунок 3).
Таблица 4
Использование данных ШГР как предиктора клинического диагноза СГХС в условиях отсутствия патогенных ВНП в генах LDLR, APOB и PCSK9
|
ШГР |
Фенотип |
AUC |
р |
|
Willer CJ, et al. (2013) [21] |
ХС ЛНП |
0,66 |
1,49×10⁻⁹ |
|
Общий ХС |
0,76 |
2,34×10⁻²⁷ |
|
|
Selvaraj MS, et al. (2022) [22] |
ХС ЛНП |
0,70 |
1,01×10⁻¹⁵ |
|
Общий ХС |
0,69 |
1,61×10⁻¹⁴ |
|
|
Xu Y, et al. (2023) [23] |
ХС ЛНП |
0,64 |
4,45×10⁻⁷ |
|
Общий ХС |
0,72 |
6,29×10⁻⁵ |
Примечание: ЛНП — липопротеины низкой плотности, СГХС — семейная гиперхолестеринемия, ХС — холестерин, ШГР — шкала генетического риска, AUC — площадь под ROC-кривой.
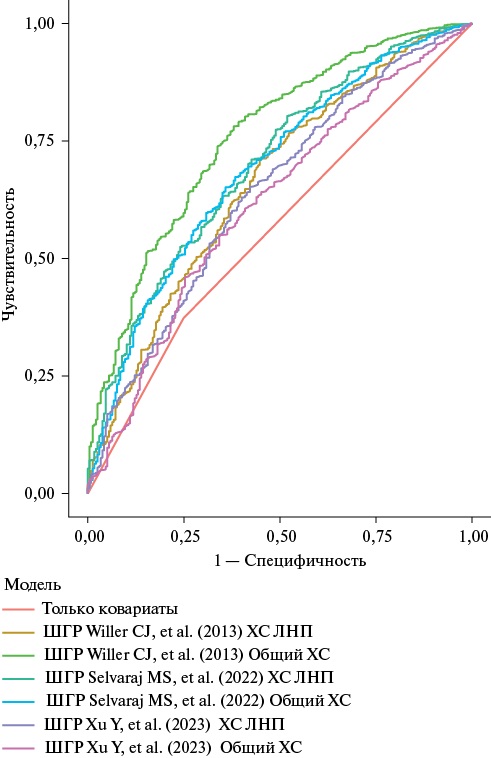
Рис. 3 ROC-кривые для моделей предсказания клинического диагноза СГХС среди неносителей патогенных вариантов с учетом ШГР Willer CJ, et al. (2013) [21], Selvaraj MS, et al. (2022) [22], Xu Y, et al. (2023) [23].
Примечание: ЛНП — липопротеины низкой плотности, СГХС — семейная гиперхолестеринемия, ХС — холестерин, ШГР — шкала генетического риска. Цветное изображение доступно в электронной версии журнала.
Обсуждение
В настоящем исследовании оценены применимость ШГР, связанных с уровнями общего ХС и ХС ЛНП, к российской популяции, а также возможность использования результатов расчета ШГР для предсказания наличия направительного диагноза гетерозиготной СГХС, обусловленной полигенной ГХС.
Вопрос применимости ШГР, разработанных на популяции, отличной от исследуемой, до сих пор остается открытым [24]. В настоящем исследовании была проведена оценка применимости 6 различных ШГР для популяционных данных из Ивановской области (ЭССЕ-Иваново) основываясь на показателе R². Значения R² не превышали 6,23% для ХС ЛНП и 5,98% для общего ХС. Данные результаты оказались несколько ниже, чем заявленные авторами двух шкал [23] и чем данные из литературы [25]. Этот факт связан как с тем, что ШГР применялись к популяции, отличной от той, на которой их разрабатывали (европейская [21][23] или мультиэтническая [22]), так и с тем, что в случаях шкал Selvaraj MS, et al. (2022) [22] и Xu Y, et al. (2023) [23] в выборке была идентифицирована лишь часть из ВНП из оригинальных исследований (от 9 до 18%).
Для трех групп в исследовании — популяционная выборка ЭССЕ-Иваново, пациенты с клиническим диагнозом СГХС носители и неносители патогенных ВНП в генах LDLR, APOB и PCSK9 — было показано значимое различие в значениях всех исследуемых ШГР. В рамках исследования на российской выборке удалось подтвердить выводы работы Wu H, et al. [12]: четыре из шести ШГР значимо различаются по распределению значений между носителями и неносителями патогенных ВНП в генах LDLR, APOB и PCSK9. Важным дополнением нашей работы к исследованию Wu H, et al. (2021) [12] является использование ШГР для общего ХС, помимо ШГР для ХС ЛНП. В рамках работы было показано, что для пациентов с выявленным патогенным ВНП наблюдаются как более высокие значения ХС ЛНП и общего ХС, так и более высокая частота наличия ксантом и липоидных дуг, чем среди пациентов без выявленного ВНП. Данные находки подтверждают результаты работы Khan TZ, et al. (2020) [26]. Отсутствие значимых различий в частоте случаев ИБС может быть объяснено тем, что выборка пациентов с патогенным ВНП была значимо младше выборки пациентов без патогенного ВНП. Ограничением настоящего исследования является невозможность оценить влияние классических факторов риска сердечно-сосудистых событий на клиническое течение СГХС ввиду отсутствия данных [27]. Более того, было показано, что данные ШГР позволяют построить предиктор направительного диагноза гетерозиготная СГХС, обусловленного полигенной ГХС, что может быть полезно для более точной оценки сердечно-сосудистого риска у таких пациентов. Известно, что в мировой практике данные ШГР часто используют для постановки диагноза "полигенная ГХС" среди людей с клиническим диагнозом СГХС, у которых патогенные ВНП не выявлены [28]. В настоящей работе удалось получить модели для предсказания направительного клинического диагноза гетерозиготная СГХС, схожие по эффективности с описанными в литературе [29].
Заключение
Впервые в России проведено исследование применимости ШГР для общего ХС и ХС ЛНП к пациентам с клиническим диагнозом СГХС. Показано, что данные ШГР могут быть использованы для дифференциальной диагностики у пациентов с клиническим диагнозом гетерозиготной СГХС и более точной оценки сердечно-сосудистого риска. Результаты исследования подчеркивают необходимость дальнейшей разработки собственных ШГР для российской популяции, которые бы позволили достичь лучших показателей качества, чем ШГР зарубежных авторов.
Отношения и деятельность. Работа выполнена при поддержке Государственного задания "Разработка модели предсказания пенетрантности и экспрессивности причинных вариантов наследственных моногенных заболеваний сердечно-сосудистой системы".
Список литературы
1. Ежов М. В., Кухарчук В. В., Сергиенко И. В. и др. Нарушения липидного обмена. Клинические рекомендации 2023. Российский кардиологический журнал. 2023;28(5):5471. doi:10.15829/1560-4071-2023-5471.
2. Meshkov A, Ershova A, Kiseleva A, et al. The LDLR, APOB, and PCSK9 Variants of Index Patients with Familial Hypercholesterolemia in Russia. Genes. 2021;12(1):66. doi:10.3390/genes12010066.
3. Meshkov AN, Ershova AI, Kiseleva AV, et al. The Prevalence of Heterozygous Familial Hypercholesterolemia in Selected Regions of the Russian Federation: The FH-ESSE-RF Study. J Pers Med. 2021;11(6):464. doi:10.3390/jpm11060464.
4. Wang J, Dron JS, Ban MR, et al. Polygenic versus monogenic causes of hypercholesterolemia ascertained clinically. Arterioscler Thromb Vasc Biol. 2016;36(12):2439-45. doi:10.1161/ATVBAHA.116.308027.
5. Tandirerung FJ. The Clinical Importance of Differentiating Monogenic Familial Hypercholesterolemia from Polygenic Hypercholesterolemia. Curr Cardiol Rep. 2022;24(11):1669-77. doi:10.1007/s11886-022-01783-5.
6. Trinder M, Francis GA, Brunham LR. Association of Monogenic vs Polygenic Hypercholesterolemia With Risk of Atherosclerotic Cardiovascular Disease. JAMA Cardiol. 2020;5(4):390-9. doi:10.1001/jamacardio.2019.5954.
7. Лимонова А. С., Ершова А. И., Киселева А. В. и др. Валидация шкал генетического риска развития артериальной гипертензии на популяции региона Центральной России. Кардиоваскулярная терапия и профилактика. 2023;22(12): 3801. doi:10.15829/1728-8800-2023-3801.
8. Зайченока М., Ершова А. И., Киселева А. В. и др. Поиск и репликация ассоциаций вариантов генома с уровнями липидов в выборке из представителей российской популяции. Кардиоваскулярная терапия и профилактика. 2023;22(12):3871. doi:10.15829/1728-8800-2023-3871.
9. Киселева А. В., Сопленкова А. Г., Куценко В. А. и др. Валидация шкал генетического риска ожирения на выборке населения регионов России. Кардиоваскулярная терапия и профилактика. 2023;22(10):3755. doi:10.15829/1728-8800-2023-3755.
10. Trinder M, Paquette M, Cermakova L, et al. Polygenic Contribution to Low-Density Lipoprotein Cholesterol Levels and Cardiovascular Risk in Monogenic Familial Hypercholesterolemia. Circ-Genom Precis Med. 2020;13(5):515-23. doi:10.1161/CIRCGEN.120.002919.
11. Khera AV, Chaffin M, Aragam KG, et al. Genome-wide polygenic scores for common diseases identify individuals with risk equivalent to monogenic mutations. Nat Genet. 2018;50(9):1219-24. doi:10.1038/s41588-018-0183-z.
12. Wu H, Forgetta V, Zhou S, et al. Polygenic Risk Score for LowDensity Lipoprotein Cholesterol Is Associated With Risk of Ischemic Heart Disease and Enriches for Individuals With Familial Hypercholesterolemia. Circ-Genom Precis Med. 2021;14(1): e003106. doi:10.1161/CIRCGEN.120.003106.
13. Ramensky VE, Ershova AI, Zaicenoka M, et al. Targeted sequencing of 242 clinically important genes in the Russian population from the ivanovo region. Front Genet. 2021;12:709419. doi:10.3389/fgene.2021.709419.
14. Чубыкина У. В., Ежов М. В., Рожкова Т. А. и др. Пятилетний период наблюдения за пациентами с гомои гетерозиготной семейной гиперхолестеринемией в регистре РЕНЕССАНС. Атеросклероз и дислипидемии. 2023;1(50):0001. doi:10.34687/2219-8202.JAD.2023.01.0001.
15. Покровская М. С., Борисова А. Л., Метельская В. А. и др. Роль биобанкирования в организации крупномасштабных эпидемиологических исследований. Кардиоваскулярная терапия и профилактика. 2021;20(5):2958. doi:10.15829/17288800-2021-2958.
16. Копылова О. В., Ершова А. И., Покровская М. С. и др. Популяционно-нозологический исследовательский биобанк "НМИЦ ТПМ": анализ коллекций биообразцов, принципы сбора и хранения информации. Кардиоваскулярная терапия и профилактика. 2021;20(8):3119. doi:10.15829/1728-8800-2021-3119.
17. Poplin R, Ruano-Rubio V, DePristo MA, et al. Scaling accurate genetic variant discovery to tens of thousands of samples. bioRxiv. 2017:201178. doi:10.1101/201178.
18. McLaren W, Gil L, Hunt SE, et al. The Ensembl Variant Effect Predictor. Genome Biol. 2016;17(1):122. doi:10.1186/s13059-0160974-4.
19. Landrum MJ, Lee JM, Riley GR, et al. ClinVar: public archive of relationships among sequence variation and human phenotype. Nucleic Acids Res. 2014;42(1):D980-5. doi:10.1093/nar/gkt1113.
20. Purcell S, Neale B, Todd-Brown K, et al. PLINK: a tool set for wholegenome association and population-based linkage analyses. Am J Hum Genet. 2007;81(3):559-75. doi:10.1086/519795.
21. Willer CJ, Schmidt EM, Sengupta S, et al. Discovery and refinement of loci associated with lipid levels. Nat Genet. 2013;45(11):1274-83. doi:10.1038/ng.2797.
22. Selvaraj MS, Li X, Li Z, et al. Whole genome sequence analysis of blood lipid levels in >66,000 individuals. Nat Commun. 2022;13(1). doi:10.1038/s41467-022-33510-7.
23. Xu Y, Ritchie SC, Liang Y, et al. An atlas of genetic scores to predict multi-omic traits. Nature. 2023;616(7955):123-31. doi:10.1038/s41586-023-05844-9.
24. Albert EA, Kondratieva OA, Baranova EE, et al. Transferability of the PRS estimates for height and BMI obtained from the European ethnic groups to the Western Russian populations. Front Genet. 2023;14:1086709. doi:10.3389/fgene.2023.1086709.
25. Leal LG, Hoggart C, Jarvelin MR, et al. A polygenic biomarker to identify patients with severe hypercholesterolemia of polygenic origin. Mol Genet. 2020;8(6):e1248. doi:10.1002/mgg3.1248.
26. Khan TZ, Breen J, Neves E, et al. Prevalence of cardiovascular events in genetically confirmed versus unconfirmed familial hypercholesterolaemia. Glob Cardiol Sci Pract. 2020;2020(2): e202024. doi:10.21542/gcsp.2020.24.
27. Pérez de Isla L, Alonso R, Mata N, et al. Predicting Cardiovascular Events in Familial Hypercholesterolemia: The SAFEHEART Registry (Spanish Familial Hypercholesterolemia Cohort Study). Circulation. 2017;135(22):2133-44. doi:10.1161/CIRCULATIONAHA.116.024541.
28. Cupido AJ, Tromp TR, Hovingh GK. The clinical applicability of polygenic risk scores for LDL-cholesterol: considerations, current evidence and future perspectives. Curr Opin Lipidol. 2021; 32(2):112-6. doi:10.1097/MOL.0000000000000741.
29. Vanhoye X, Bardel C, Rimbert A, et al. A new 165-SNP lowdensity lipoprotein cholesterol polygenic risk score based on next generation sequencing outperforms previously published scores in routine diagnostics of familial hypercholesterolemia. Transl Res. 2023;255:119-27. doi:10.1016/j.trsl.2022.12.002.
Об авторах
М. ЗайченокаЛатвия
Аспирант Физтех-школы биологической и медицинской физики, аспирант лаборатории геномной и медицинской биоинформатики Института персонализированной терапии и профилактики.
Долгопрудный, Москва
А. Н. Мешков
Россия
д.м.н., руководитель Института персонализированной терапии и профилактики.
Москва
А. В. Киселева
Россия
к.б.н., руководитель лаборатории молекулярной генетики в.н.с.
Москва
А. И. Ершова
Россия
д.м.н., руководитель лаборатории клиномики, зам. директора по фундаментальной науке.
Москва
Е. А. Сотникова
Россия
с.н.с. лаборатории молекулярной генетики.
Москва
А. А. Жарикова
Россия
к.б.н., в.н.с. лаборатории молекулярной генетики, старший преподаватель факультета биоинженерии и биоинформатики.
Москва
Ю. В. Вяткин
Россия
программист лаборатории геномной и медицинской биоинформатики Института персонализированной терапии и профилактики, с.н.с.
Москва
В. И. Михайлина
Россия
м.н.с. отдела персонализированной диагностики, терапии и профилактики атеросклеротических сердечно-сосудистых заболеваний Института персонализированной терапии и профилактики.
Москва
А. А. Букаева
Россия
н.с. лаборатории клиномики.
Москва
М. С. Покровская
Россия
к.б.н., в.н.с. лаборатории "Банк биологического материала" Института персонализированной терапии и профилактики.
Москва
В. Е. Раменский
Россия
к.ф.м.н., в.н.с., руководитель лаборатории геномной и медицинской биоинформатики Института персонализированной терапии и профилактики, доцент факультета биоинженерии и биоинформатики, руководитель научной группы "ИИ в биоинформатике и медицине" Института перспективных исследований проблем искусственного интеллекта и интеллектуальных систем.
Москва
О. М. Драпкина
Россия
д.м.н., профессор, академик РАН, директор.
Москва
Дополнительные файлы
Что известно о предмете исследования?
- Шкалы генетического риска (ШГР) позволяют оценить вклад в развитие фенотипа вариантов с малым эффектом.
- ШГР для холестерина (ХС) липопротеинов низкой плотности и общего ХС предположительно могут быть использованы для дифференциальный диагностики больных с гетерозиготной семейной гиперхолестеринемией (СГХС).
Что добавляют результаты исследования?
- Распределения ШГР для уровней ХС липопротеинов низкой плотности и общего ХС значимо различаются для среднестатистической популяции и выборки пациентов с гетерозиготной СГХС.
- Значения ШГР позволяют предсказывать наличие клинического диагноза "гетерозиготная СГХС" среди неносителей патогенных вариантов.
Рецензия
Для цитирования:
Зайченока М., Мешков А.Н., Киселева А.В., Ершова А.И., Сотникова Е.А., Жарикова А.А., Вяткин Ю.В., Михайлина В.И., Букаева А.А., Покровская М.С., Раменский В.Е., Драпкина О.М. Использование шкал генетического риска для дифференциальной диагностики у лиц с клиническим диагнозом семейная гиперхолестеринемия. Кардиоваскулярная терапия и профилактика. 2024;23(12):4251. https://doi.org/10.15829/1728-8800-2024-4251. EDN: UYQMBM
For citation:
Zaychenoka M., Meshkov A.N., Kiseleva A.V., Ershova A.I., Sotnikova E.A., Zharikova A.A., Vyatkin Yu.V., Mikhailina V.I., Bukaeva A.A., Pokrovskaya M.S., Ramenskiy V.E., Drapkina O.M. Use of polygenic risk scores for differential diagnostics for patients with clinical diagnosis of familial hypercholesterolemia. Cardiovascular Therapy and Prevention. 2024;23(12):4251. (In Russ.) https://doi.org/10.15829/1728-8800-2024-4251. EDN: UYQMBM
JATS XML

























































