Перейти к:
Поиск и репликация ассоциаций вариантов генома с уровнями липидов в выборке из представителей российской популяции
https://doi.org/10.15829/1728-8800-2023-3871
EDN: GTFBBS
Аннотация
Цель. Провести поиск ассоциаций для показателей липидного профиля (уровня холестерина липопротеинов низкой и высокой плотности, триглицеридов и общего холестерина) на популяционных выборках из двух регионов Российской Федерации и репликационный анализ ранее опубликованного полногеномного поиска ассоциаций (англ. genome-wide association studies, GWA study, GWAS) для жителей трех других регионов Российской Федерации.
Материал и методы. В исследование включены репрезентативные выборки из Вологодской (n=689) и Ивановской (n=1675) областей, собранных для исследования ЭССЕ-РФ (Эпидемиология сердечно-сосудистых заболеваний в регионах Российской Федерации). Участникам исследования проводили определение показателей липидного профиля и таргетное секвенирование. Модель линейной регрессии с учетом пола, возраста и приема статинов была использована для оценки ассоциаций геномных вариантов с показателями липидного спектра. В работе проведена репликация результатов исследования Usoltsev D, et al., 2023, выполненного на популяционных выборках лиц из Санкт-Петербурга, Оренбургской и Самарской областей.
Результаты. При поиске ассоциаций были идентифицированы варианты, для которых ранее уже были выявлены ассоциации с липидными показателями на выборке из представителей российской популяции. Доля реплицированных вариантов составила 89% для выборки из Вологодской области и 92% для выборки из Ивановской области. Направления эффектов всех реплицированных вариантов в ранее опубликованном исследовании (выборки из Оренбургской и Самарской областей и Санкт-Петербурга) и в обеих исследуемых выборках (выборки из Ивановской и Вологодской областей) совпадают.
Заключение. Результаты поиска ассоциаций с липидными показателями на разных российских выборках согласуются между собой.
Для цитирования:
Зайченока М., Ершова А.И., Киселева А.В., Сотникова Е.А., Вяткин Ю.В., Жарикова А.А., Покровская М.С., Шальнова С.А., Раменский В.Е., Мешков А.Н., Драпкина О.М. Поиск и репликация ассоциаций вариантов генома с уровнями липидов в выборке из представителей российской популяции. Кардиоваскулярная терапия и профилактика. 2023;22(12):3871. https://doi.org/10.15829/1728-8800-2023-3871. EDN: GTFBBS
For citation:
Zaichenoka M., Ershova A.I., Kiseleva A.V., Sotnikova E.A., Vyatkin Yu.V., Zharikova А.A., Pokrovskaya M.S., Shalnova S.A., Ramensky V.E., Meshkov A.N., Drapkina O.M. Search and replication of associations of genome variants with lipid levels in a Russian sample. Cardiovascular Therapy and Prevention. 2023;22(12):3871. (In Russ.) https://doi.org/10.15829/1728-8800-2023-3871. EDN: GTFBBS
Введение
Основной причиной смерти как в России, так и во всем мире остаются сердечно-сосудистые заболевания, среди которых ведущее место занимают заболевания атеросклеротического генеза [1]. Одним из ключевых факторов риска развития атеросклероза является гиперлипидемия. Уровень циркулирующих липидов зависит от образа жизни, сопутствующих заболеваний и генетических факторов [2].
Выявлению независимых ассоциаций между вариантами нуклеотидной последовательности (ВНП) и уровнями циркулирующих липидов служит такой метод исследований, как полногеномный поиск ассоциаций (GWAS). Результаты GWAS используются для создания шкал генетического риска комплексных заболеваний, представляющих собой оценку полигенного вклада в развитие признака или заболевания [3]. Полигенные шкалы риска имеют высокий потенциал применения в клинической практике для доклинической стратификации риска [4].
При проведении GWAS на выборке из 1,65 млн человек (чел.) разного происхождения идентифицирован 941 ВНП, ассоциированный с уровнями общего холестерина (ХС), ХС липопротеинов низкой плотности (ЛНП), ХС липопротеинов высокой плотности (ЛВП) и триглицеридов (ТГ) [5]. В другом исследовании, включившем 312571 участников, авторы подтвердили 188 уже известных ассоциаций с уровнями липидов, а также показали 118 новых ассоциаций [6]. У представителей российской популяции ранее был проведен только один GWAS на выборках, состоящих из лиц, проживающих в Санкт-Петербурге, Самарской и Оренбургской областях [7]. В этом исследовании были идентифицированы 30 ВНП, значимо ассоциированных с липидными показателями [7]. Подобные исследования нуждаются в репликации результатов на независимых выборках для подтверждения найденных ассоциаций [8], что в исходной статье не было сделано.
Цель настоящей работы — провести поиск ассоциаций для четырех показателей липидного профиля (общий ХС, ХС ЛНП, ХС ЛВП, ТГ и) на популяционных выборках из двух регионов Российской Федерации (Вологодской и Ивановской областей) для поиска возможных новых ассоциаций и репликационный анализ ранее опубликованного GWAS [7], полученного на выборке из жителей Санкт-Петербурга, Самарской и Оренбургской областей.
Материал и методы
Выборки. В исследование вошли 2364 человека в возрасте 21-67 лет из эпидемиологического исследования ЭССЕ-РФ (Эпидемиология сердечно-сосудистых заболеваний в регионах России): 689 чел. из Вологодской области (участники ЭССЕ-Вологда) [9] и 1675 чел. из Ивановской области (участники ЭССЕ-Иваново) [10]. Включение участников исследования проводилось случайным образом согласно методу Киша. В анализ были включены демографические (пол, возраст) и клинические (прием статинов) данные участников исследования, полученные во время анкетирования в рамках ЭССЕ-Иваново и ЭССЕ-Вологда.
Сбор и хранение биообразцов выполняли согласно регламенту биобанкирования в Биобанке ФГБУ "НМИЦ ТПМ" Минздрава России (г. Москва) [11][12]. У всех участников определяли уровни общего ХС, ХС ЛНП, ХС ЛВП и ТГ в сертифицированной лаборатории с помощью автоматического биохимического анализатора крови ARCHITECT c8000 и диагностических наборов (Abbott, США).
Исследование одобрено Независимым Этическим Комитетом ФГБУ "НМИЦ ТПМ" Минздрава России (номер протокола 07-03/12 от 03.07.2012). Все участники дали письменное информированное согласие.
Генетический анализ. Для выделения ДНК из цельной крови использовали набор QIAamp DNA Blood Mini Kit (Qiagen, Германия). Оценку качества и определение концентрации выделенной дезоксирибонуклеиновой кислоты (ДНК) проводили с помощью приборов NanoDrop OneC (Thermo Fisher Scientific, США) и Qubit 4.0 (Thermo Fisher Scientific, США). Подготовка библиотек для секвенирования была проведена с помощью набора SeqCap EZ Prime Choice Library (Roche, Швейцария).
Для каждого участника исследования было проведено секвенирование нового поколения с использованием одной из двух таргетных панелей вариантов, разработанных и используемых нами для диагностики и определения предрасположенности к сердечно-сосудистым и прочим мультифакторным заболеваниям. Панель, использованная для участников выборки ЭССЕ-Вологда, включала 217 генов и дополнительно 18489 ВНП. Панель, использованная для участников выборки ЭССЕ-Иваново, включала 242 гена и 2042 ВНП [11]. Секвенирование было проведено на приборе NextSeq 550 (Illumina, США) с получением парноконцевых прочтений длиной 75 или 150 п.н. Все этапы секвенирования были проведены в соответствии с протоколами производителей.
Биоинформатический анализ. Парноконцевые прочтения в формате fastq были выровнены на референсный геном GRCh38. Обработка данных и оценка контроля качества выполнялись с помощью специально разработанного пайплайна [10] на базе GATK 3.8 [13]. Аннотацию однонуклеотидных ВНП и коротких инсерций и делеций осуществляли с помощью ENSEMBL Variant Effect Predictor (v. 100) [14] и базы данных dbSNP [15].
Результаты GWAS Usoltsev D, et al. (2023) [7], репликация которого представлена в этой работе, были скачаны в виде сводных статистик с координатами, соответствующими сборке генома GRCh37. Для дальнейшей работы координаты ВНП были переведены в соответствие со сборкой генома GRCh38 с помощью программы LiftOver [16]. В качестве ВНП, значимо ассоциированных с исследуемыми фенотипами, рассматривали варианты, для которых p-значение <5×10-8.
Статистический анализ. Статистический анализ проводили с использованием библиотек hail v. 0.2.105-acd89e80c3451 и scipy v. 1.2.1 [17] языка программирования Python v. 3.7.42. Значения ТГ были логарифмированы (logТГ), т.к. изначальное распределение было асимметрично. Непрерывные данные представлены в виде медианы и интерквартильного размаха (Me [Q25; Q75]). Для сравнения качественных параметров использовали критерий χ2 с поправкой Йейтса. Сравнение распределений количественных параметров в независимых выборках проводили с использованием критерия Манна-Уитни. Для расчета корреляций использовался коэффициент корреляции Спирмена.
Для поиска ассоциаций и репликации результатов GWAS [7] были отобраны ВНП, генотипы которых определены у >90% исследуемых в каждой выборке, с количеством аллельных копий не <10 и находящиеся в равновесии Харди-Вайнберга (p-значение >10-4). Модель линейной регрессии с учетом пола, возраста и приема статинов была использована для оценки ассоциаций ВНП с общим ХС, ХС ЛНП, ХС ЛВП и logТГ. Порог значимости ассоциаций выбирали с учетом поправки Бонферрони на множественную проверку гипотез: 0,05/k, где k — количество вариантов в соответствующей выборке. ВНП считали номинально реплицированными, если в исходном исследовании p-значение не превышало 5×10-8, а в репликационном анализе для этого же ВНП получено p-значение не >0,05 [18], и реплицированными, если в исходном исследовании p-значение <5×10-8, а в репликационном анализе для этого же ВНП получено p-значение <0,05/k, где k — количество значимых в исходном исследовании ВНП для соответствующих липидных показателей (общий ХС, ХС ЛНП, ХС ЛВП, logТГ), найденных в выборке (ЭССЕ-Вологда или ЭССЕ-Иваново). Подробная схема исследования представлена на рисунке 1.
Результаты
Клиническая характеристика участников исследования представлена в таблице 1. Участники ЭССЕ-Вологда отличались достоверно более высоким уровнем ХС ЛНП и более низкими уровнями ХС ЛВП и ТГ по сравнению с лицами из ЭССЕ-Иваново.
Поиск ассоциаций
В выборке ЭССЕ-Иваново было выявлено 25124 ВНП, прошедших контроль качества, а в выборке ЭССЕ-Вологда — 95656 ВНП. Варианты считали значимо ассоциированными, если р-значение не превышало 5,23×10-7 в случае выборки ЭССЕ-Вологда (0,05/95656 вариантов) и 1,99×10-6 в случае выборки ЭССЕ-Иваново (0,05/25124 вариантов).
Результаты поиска ассоциаций с общим ХС, ХС ЛНП, ХС ЛВП и logТГ представлены на рисунках 2-7 и в таблицах 2-6. В случае ХС ЛНП 3 ВНП — rs7412, rs445925, rs72654473 — показали значимые ассоциации в обеих выборках (таблица 2, рисунки 2 и 6). При поиске ассоциаций с ХС ЛВП было выявлено 16 ВНП, ассоциированных с признаком, при исследовании выборки ЭССЕ-Иваново и 5 ВНП — выборки ЭССЕ-Вологда (таблицы 3-4, рисунки 3 и 7). При поиске ассоциаций с logТГ и общим ХС на выборке ЭССЕ-Вологда значимых ассоциаций выявлено не было. При исследовании выборки ЭССЕ-Иваново выявлен один ВНП, значимо ассоциированный с общим ХС (rs7412, таблица 6, рисунок 4), и 7 ВНП — с logТГ (таблица 5, рисунок 5).
Репликация результатов GWAS
В сводных статистиках в работе Usoltsev D, et al. (2023) [7] для 4 исследуемых липидных показателей суммарно были представлены данные по 7552009 ВНП. Чтобы убедиться в отсутствии значимых генетических различий между анализируемыми выборками, были сопоставлены частоты ВНП, идентифицированные в исследуемых когортах ЭССЕ (14542 ВНП в когорте ЭССЕ-Иваново и 68245 ВНП в ЭССЕ-Вологда) и когорте Usoltsev D, et al. (2023) [7]. Корреляции между частотами ВНП, найденных в выборках ЭССЕ-Вологда и ЭССЕ-Иваново, и частотами в работе Usoltsev D, et al. (2023) [7] составили 99,5 и 99,8%, соответственно.
В исследовании Usoltsev D, et al. (2023) [7] было обнаружено 30 ВНП, статистически значимо ассоциированных с ХС ЛНП, 25 ВНП — с ХС ЛВП, 1 ВНП — с logТГ и 22 ВНП — с общим ХС.
Из них в выборках ЭССЕ-Вологда и ЭССЕ-Иваново было выявлено соответственно 53 и 43% ВНП, ассоциированных с уровнем ХС ЛНП, 60% и 36% — с ХС ЛВП, 18 и 13% — с общим ХС. Единственный ВНП, ассоциированный с уровнем ТГ, был выявлен в обеих выборках (таблица 7). После применения поправки пороговые уровни p-значений для репликации составили 1,25×10-2 для общего ХС, 3,13×10-3 для ХС ЛНП, 3,33×10-3 для ХС ЛВП и 0,05 для logТГ в случае выборки ЭССЕ-Вологда и 1,66×10-2 для общего ХС, 3,84×10-3 для ХС ЛНП, 5,55×10-3 для ХС ЛВП и 0,05 для logТГ, в случае выборки ЭССЕ-Иваново.
Результаты репликации представлены в таблицах 8-11. В случае ХС ЛНП в выборке ЭССЕ-Вологда были номинально реплицированы все ВНП, кроме rs4970836, и все ВНП в выборке ЭССЕ-Иваново, кроме rs4970834. При применении поправки на количество ВНП реплицированными в случае ЭССЕ-Вологда оказались 7 ВНП (rs7412, rs1160983, rs72654473, rs445925, rs429358, rs56131196, rs4420638), в случае ЭССЕ-Иваново — 6 ВНП (rs7412, rrs72654473, rs445925, rs429358, rs56131196, rs4420638). Для ХС ЛВП и logТГ были реплицированы все идентифицированные в выборках ВНП. Для уровня общего ХС в выборке ЭССЕ-Вологда был реплицирован только один ВНП — rs7412, в выборке ЭССЕ-Иваново — rs7412 и rs1367117. Общая доля реплицированных ВНП составила 89% для выборки ЭССЕ-Вологда и 92% для ЭССЕ-Иваново.
Все β-оценки реплицированных ВНП совпадали по направлению эффекта ВНП с заявленным в работе Usoltsev D, et al. (2023) [7].
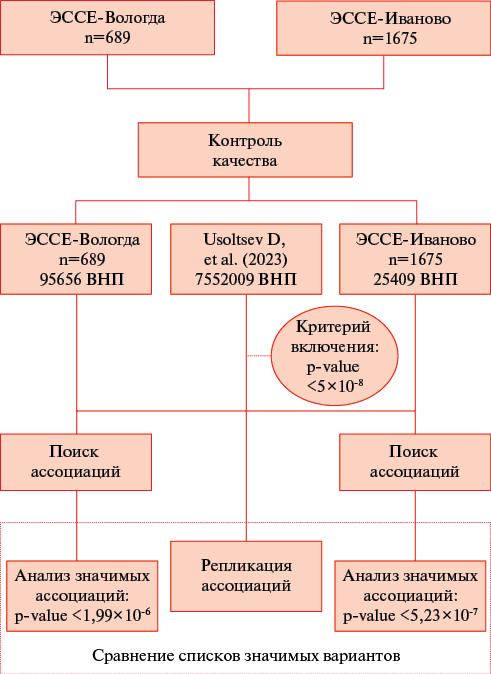
Рис. 1 Схема исследования.
Примечание: ВНП — варианты нуклеотидной последовательности.
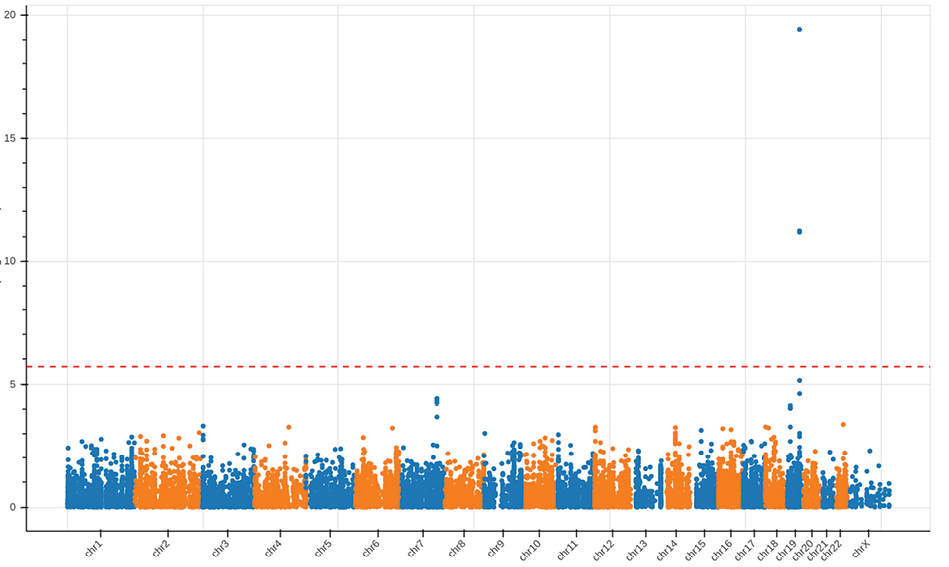
Рис. 2 Манхэттенский график для результатов поиска ассоциаций с ХС ЛНП в выборке ЭССЕ-Иваново.
Примечание: ХС ЛНП — холестерин липопротеинов низкой плотности.
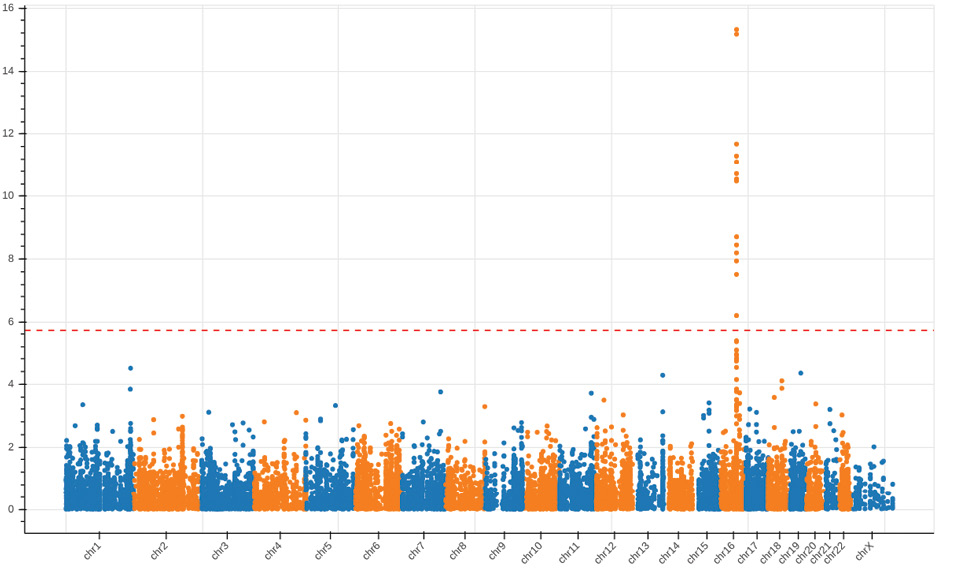
Рис. 3 Манхэттенский график для результатов поиска ассоциаций с ХС ЛВП в выборке ЭССЕ-Иваново.
Примечание: ХС ЛВП — холестерин липопротеинов высокой плотности.
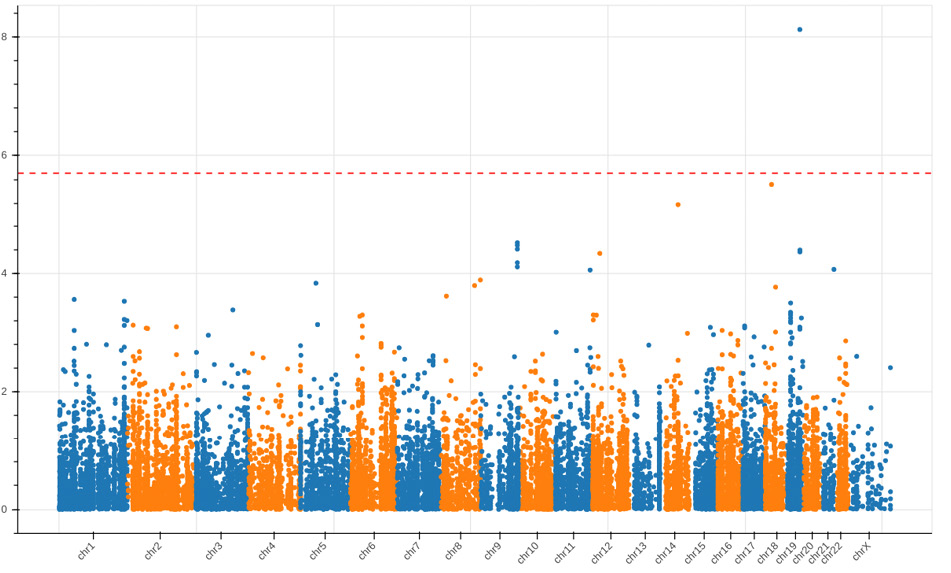
Рис. 4 Манхэттенский график для результатов поиска ассоциаций с ОХС в выборке ЭССЕ-Иваново.
Примечание: ХС — холестерин.
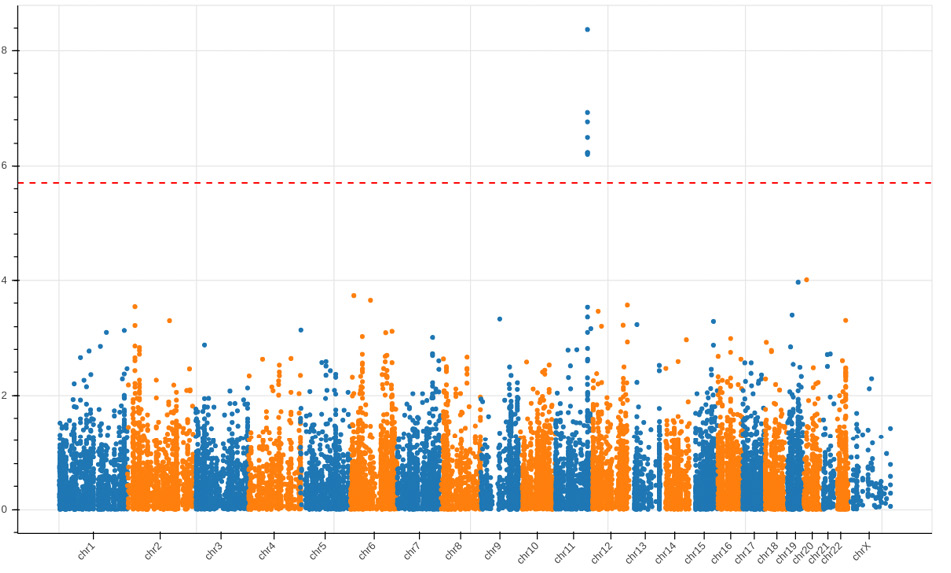
Рис. 5 Манхэттенский график для результатов поиска ассоциаций с logТГ в выборке ЭССЕ-Иваново.
Примечание: logТГ — логарифм уровня триглицеридов.
Таблица 1
Клиническая характеристика участников исследования
ЭССЕ-Вологда, n=689 | ЭССЕ-Иваново, n=1675 | p | |
Мужской пол, n (%) | Мужчин: 313 (45,4) | Мужчин: 624 (37,3) | <0,001 |
Возраст, лет (Ме [Q25; Q75]) | 47 [ 35; 55] | 49 [ 39; 56] | <0,001 |
Курящие, n (%) | 163 (23,7%) | 605 (36,1%) | <0,001 |
ХС ЛНП, ммоль/л (Ме [Q25; Q75]) | 3,34 [ 2,68; 4,05] | 3,22 [ 2,42; 4,01] | 0,004 |
ХС ЛВП, ммоль/л (Ме [Q25; Q75]) | 1,24 [ 1,05; 1,45] | 1,38 [ 1,18; 1,62] | <0,001 |
ТГ, ммоль/л (Ме [Q25; Q75]) | 1,09 [ 0,75; 1,46] | 1,23 [ 0,87; 1,82] | <0,001 |
Общий ХС, ммоль/л (Ме [Q25; Q75]) | 5,12 [ 4,39; 5,96] | 5,46 [ 4,72; 6,28] | <0,001 |
Примечание: Ме — медиана, Q — квартиль, ЛВП — липопротеины высокой плотности, ЛНП — липопротеины низкой плотности, ХС — холестерин, ТГ — триглицериды. p-значения рассчитывались на основе критерия Манна-Уитни или χ2 с поправкой Йейтса и отражают различие между выборками.
Таблица 2
Значимые варианты, выявленные при поиске ассоциаций с ХС ЛНП на выборках ЭССЕ-Вологда и ЭССЕ-Иваново
ЭССЕ-Вологда | ЭССЕ-Иваново | ||||
Вариант | Ген | р-значение | β-оценка | р-значение | β-оценка |
rs7412 | APOE | 3,94×10-11 | -0,60 | 3,73×10-20 | -0,61 |
rs445925 | APOE | 8,33×10-9 | -0,46 | 5,96×10-12 | -0,42 |
rs72654473 | APOE | 3,48×10-9 | -0,48 | 6,69×10-12 | -0,42 |
Примечание: ХС ЛНП — холестерин липопротеинов низкой плотности.
Таблица 3
Значимые варианты, выявленные при поиске ассоциаций с ХС ЛВП на выборке ЭССЕ-Иваново
Вариант | Ген | р-значение | β-оценка |
rs3764261 | CETP | 4,80×10-16 | 0,091 |
rs247617 | CETP | 6,75×10-16 | 0,091 |
rs34145065 | CETP | 2,07×10-12 | 0,075 |
rs711752 | CETP | 4,99×10-12 | 0,075 |
rs34620476 | CETP | 7,77×10-12 | 0,074 |
rs1532624 | CETP | 1,92×10-11 | 0,072 |
rs1532625 | CETP | 1,92×10-11 | 0,072 |
rs3816117 | CETP | 2,84×10-11 | 0,072 |
rs7205804 | CETP | 2,86×10-11 | 0,072 |
rs708272 | CETP | 3,30×10-11 | 0,070 |
rs7499892 | CETP | 1,94×10-9 | -0,085 |
rs11076175 | CETP | 3,54×10-9 | -0,084 |
rs11076176 | CETP | 6,32×10-9 | -0,085 |
rs289714 | CETP | 1,14×10-8 | 0,082 |
rs1864163 | CETP | 3,05×10-8 | -0,069 |
rs9989419 | CETP | 6,15×10-7 | 0,057 |
Примечание: ХС ЛВП — холестерин липопротеинов высокой плотности.
Таблица 4
Значимые варианты, выявленные при поиске ассоциаций с ХС ЛВП на выборке ЭССЕ-Вологда
Вариант | Ген | р-значение | β-оценка |
rs247616 | CETP | 2,37×10-7 | 0,095 |
rs3764261 | CETP | 2,68×10-7 | 0,095 |
rs247617 | CETP | 2,68×10-7 | 0,095 |
rs12149545 | CETP | 2,68×10-7 | 0,095 |
rs173539 | CETP | 3,10×10-7 | 0,094 |
Примечание: ХС ЛВП — холестерин липопротеинов высокой плотности.
Таблица 5
Значимые варианты, выявленные при поиске ассоциаций с logТГ на выборке ЭССЕ-Иваново
Вариант | Ген | р-значение | β-оценка |
rs964184 | ZPR1 | 4,28×10-9 | -0,14 |
rs10750097 | APOA5 | 1,18×10-7 | -0,11 |
rs6589565 | ZPR1 | 1,72×10-7 | -0,16 |
rs2266788 | ZPR1 | 3,20×10-7 | -0,16 |
rs662799 | ZPR1 | 5,83×10-7 | -0,16 |
rs2072560 | ZPR1 | 5,94×10-7 | -0,16 |
rs651821 | ZPR1 | 6,31×10-7 | -0,15 |
Примечание: logТГ — логарифм уровня триглицеридов.
Таблица 6
Значимый вариант, выявленный при поиске ассоциаций с общим ХС на выборке ЭССЕ-Иваново
Вариант | Ген | р-значение | β-оценка |
rs7412 | APOE | 7,46×10-9 | -0,40 |
Примечание: ХС — холестерин.
Таблица 7
Частота ВНП, значимо ассоциированных с липидными показателями в работе Usoltsev D, et al. (2023) [7], в исследуемых выборках
Репликационные выборки | Количество ВНП в выборке Usoltsev D, et al. (2023) [7] | |||
ХС ЛНП n=30 | ХС ЛВП n=25 | logТГ n=1 | Общий ХС n=22 | |
ЭССЕ-Вологда, n (%) | 16 (53) | 15 (60) | 1 (100) | 4 (18) |
ЭССЕ-Иваново, n (%) | 13 (43) | 9 (36) | 1 (100) | 3 (13) |
Примечание: ВНП — варианты нуклеотидной последовательности, ЛВП — липопротеины высокой плотности, ЛНП — липопротеины низкой плотности, ХС — холестерин, logТГ — логарифм уровня триглицеридов.
Таблица 8
Репликация значимых ВНП для ХС ЛНП
ВНП | Ген | Исследование [7] | ЭССЕ-Вологда | ЭССЕ-Иваново | |||
p-значение | β-оценка | p-значение | β-оценка | p-значение | β-оценка | ||
rs7412 | APOE | 4,70×10-29 | -0,43 | 3,94×10-11 | -0,60 | 3,73×10-20 | -0,61 |
rs1160983 | TOMM40 | 8,60×10-21 | -0,49 | 5,41×10-4 | -0,43 | – | – |
rs72654473 | APOE | 4,70×10-17 | -0,29 | 3,48×10-9 | -0,48 | 6,69×10-12 | -0,42 |
rs445925 | APOC1 | 4,30×10-16 | -0,28 | 8,33×10-9 | -0,46 | 5,96×10-12 | -0,42 |
rs429358 | APOE | 1,10×10-8 | 0,20 | 3,76×10-4 | 0,27 | 7,07×10-6 | 0,26 |
rs56131196 | APOC1 | 1,20×10-8 | 0,17 | 2,31×10-4 | 0,25 | 2,40×10-5 | 0,22 |
rs4420638 | APOC1 | 1,60×10-8 | 0,17 | 2,31×10-4 | 0,25 | 2,40×10-5 | 0,22 |
rs4970834 | CELSR2 | 1,80×10-8 | -0,15 | 1,51×10-2 | -0,16 | 0,10 | -0,08 |
rs583104 | PSRC1 | 3,30×10-8 | 0,14 | 2,93×10-2 | 0,14 | 5,88×10-3 | 0,12 |
rs4970836 | PSRC1 | 3,30×10-8 | 0,14 | 0,09 | 0,14 | – | – |
rs629301 | CELSR2 | 3,40×10-8 | 0,14 | 3,14×10-2 | 0,14 | 4,66×10-3 | 0,12 |
rs12740374 | CELSR2 | 3,70×10-8 | -0,14 | 3,56×10-2 | -0,14 | – | – |
rs646776 | CELSR2 | 3,80×10-8 | 0,14 | 3,14×10-2 | 0,14 | 4,29×10-3 | 0,13 |
rs7528419 | CELSR2 | 4,00×10-8 | -0,14 | 3,14×10-2 | -0,14 | 4,70×10-3 | -0,13 |
rs599839 | PSRC1 | 4,10×10-8 | 0,14 | 3,26×10-2 | 0,14 | 1,13×10-2 | 0,11 |
rs1277930 | PSRC1 | 4,10×10-8 | 0,14 | 1,62×10-2 | 0,15 | 6,06×10-3 | 0,12 |
Примечание: ВНП — вариант нуклеотидной последовательности, ХС ЛНП — холестерин липопротеинов низкой плотности.
Таблица 9
Репликация значимых ВНП для ХС ЛВП
ВНП | Ген | Исследование [7] | ЭССЕ-Вологда | ЭССЕ-Иваново | |||
p-значение | β-оценка | p-значение | β-оценка | p-значение | β-оценка | ||
rs247617 | CETP | 6,60×10-21 | 0,073 | 2,68×10-7 | 0,095 | 6,75×10-16 | 0,091 |
rs247616 | CETP | 1,90×10-20 | 0,072 | 2,37×10-7 | 0,095 | – | – |
rs3764261 | CETP | 2,10×10-20 | 0,072 | 2,68×10-7 | 0,095 | 4,80×10-16 | 0,091 |
rs173539 | CETP | 4,50×10-20 | 0,071 | 3,10×10-7 | 0,094 | – | – |
rs56228609 | CETP | 7,80×10-20 | 0,071 | 4,34×10-4 | 0,066 | – | – |
rs12149545 | CETP | 1,20×10-19 | 0,071 | 2,96×10-7 | 0,095 | – | – |
rs708272 | CETP | 1,20×10-13 | 0,055 | 2,86×10-5 | 0,075 | 3,30×10-11 | 0,070 |
rs711752 | CETP | 2,30×10-13 | 0,054 | 3,33×10-5 | 0,075 | 4,99×10-12 | 0,075 |
rs1800775 | CETP | 2,40×10-13 | 0,054 | 6,91×10-5 | 0,070 | – | – |
rs1532625 | CETP | 2,40×10-13 | 0,054 | 9,13×10-5 | 0,070 | 1,92×10-11 | 0,072 |
rs7205804 | CETP | 5,60×10-13 | 0,053 | 1,09×10-4 | 0,069 | 2,86×10-11 | 0,072 |
rs3816117 | CETP | 6,40×10-13 | 0,053 | 3,69×10-5 | 0,074 | 2,84×10-11 | 0,072 |
rs1532624 | CETP | 1,20×10-12 | 0,053 | 9,13×10-5 | 0,070 | 1,92×10-11 | 0,072 |
rs4783961 | CETP | 1,10×10-11 | 0,050 | 2,42×10-3 | 0,053 | – | – |
rs7499892 | CETP | 4,60×10-8 | -0,053 | 2,98×10-3 | -0,068 | 1,94×10-9 | -0,085 |
Примечание: ВНП — вариант нуклеотидной последовательности, ХС ЛВП — холестерин липопротеинов высокой плотности.
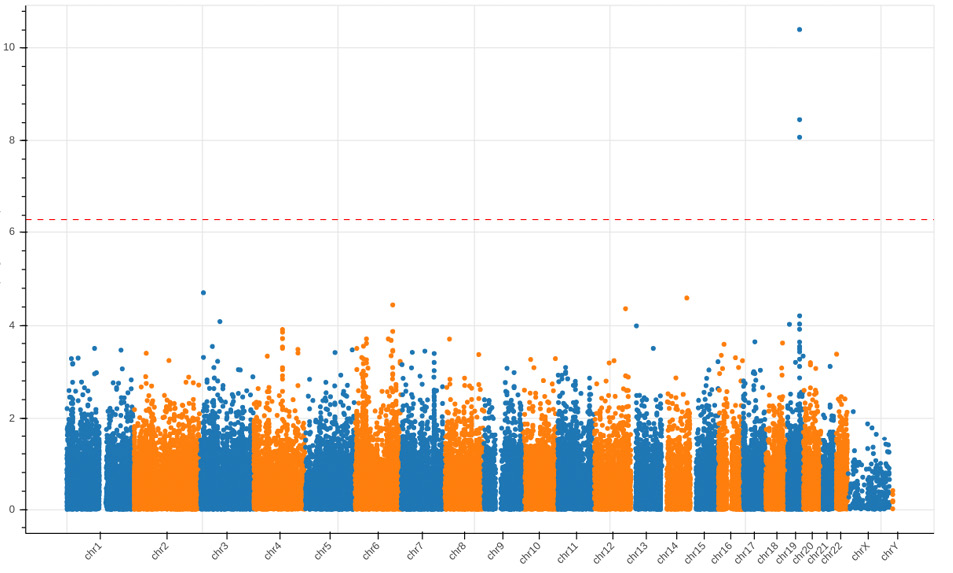
Рис. 6 Манхэттенский график для результатов поиска ассоциаций с ХС ЛНП в выборке ЭССЕ-Вологда.
Примечание: ХС ЛНП — холестерин липопротеинов низкой плотности.
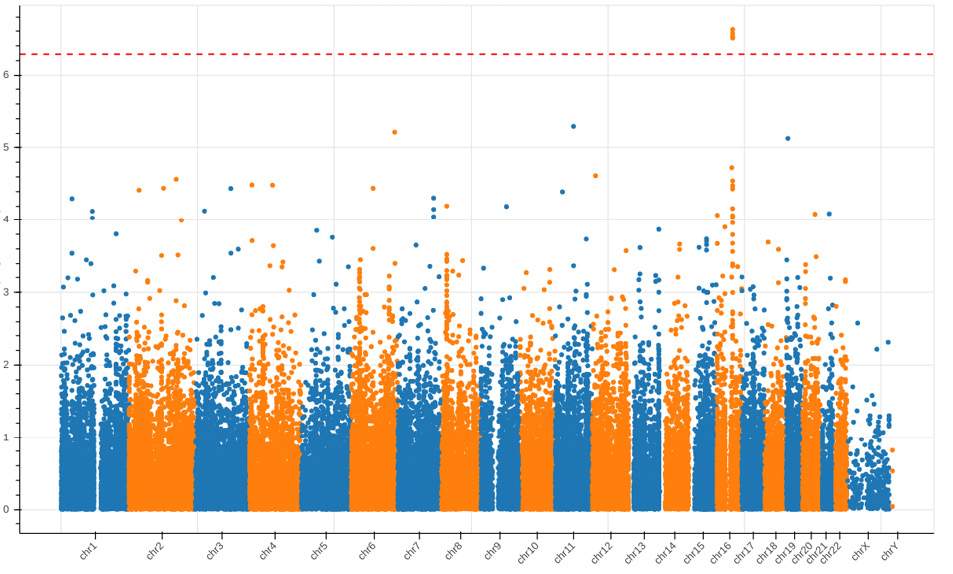
Рис. 7 Манхэттенский график для результатов поиска ассоциаций с ХС ЛВП в выборке ЭССЕ-Вологда.
Примечание: ХС ЛВП — холестерин липопротеинов высокой плотности.
Таблица 10
Репликация значимых ВНП для logТГ
ВНП | Ген | Исследование [7] | ЭССЕ-Вологда | ЭССЕ-Иваново | |||
p-значение | β-оценка | p-значение | β-оценка | p-значение | β-оценка | ||
rs964184 | ZPR1 | 1,70×10-9 | -0,11 | 1,14×10-6 | -0,19 | 4,28×10-9 | -0,14 |
Примечание: ВНП — вариант нуклеотидной последовательности, logТГ — логарифм уровня триглицеридов.
Таблица 11
Репликация значимых ВНП для общего ХС
ВНП | Ген | Исследование [7] | ЭССЕ-Вологда | ЭССЕ-Иваново | |||
p-значение | β-оценка | p-значение | β-оценка | p-значение | β-оценка | ||
rs7412 | APOE | 5,90×10-11 | -0,30 | 4,84×10-4 | -0,36 | 7,46×10-9 | -0,40 |
rs1160983 | TOMM40 | 3,30×10-9 | -0,37 | 0,20 | -0,18 | – | – |
rs4970834 | CELSR2 | 1,30×10-8 | -0,18 | 0,08 | -0,13 | 0,16 | -0,06 |
rs1367117 | APOB | 4,60×10-8 | 0,15 | 0,31 | 0,065 | 7,31×10-4 | 0,14 |
Примечание: ВНП — вариант нуклеотидной последовательности, ХС — холестерин.
Обсуждение
Проведенные на выборках ЭССЕ-Вологда и ЭССЕ-Иваново поиски ассоциаций позволили идентифицировать ВНП, статистически значимая ассоциация с липидными показателями для которых уже продемонстрирована в работе Usoltsev D, et al. (2023) [7]. Относительно небольшое количество выявленных ВНП, ассоциированных с липидными показателями, в сравнении с другими работами [19][20], вероятно, объясняется небольшими размерами выборок и ограниченным числом доступных для включения в анализ ВНП.
Полногеномные ассоциативные исследования нуждаются в репликации результатов на независимых выборках для подтверждения обнаруженных ассоциаций [7]. В данной работе была проведена репликация результатов GWAS для показателей липидного профиля Usoltsev D, el al. (2023) [7], проведенного на выборке из представителей российской популяции (из Санкт-Петербурга, Самарской и Оренбургской областей). Было показано, что 89 и 92% значимых ВНП реплицируются в выборках ЭССЕ-Вологда и ЭССЕ-Иваново номинально (см. пояснения в разделе Методы), в т.ч. не <66% с поправкой на количество ВНП. Полученные результаты хорошо согласуются с утверждением, что показатели реплицируемости GWAS достаточно высоки [7]. Более того, доля реплицированных ВНП в случае выборки ЭССЕ-Иваново больше, чем в случае выборки ЭССЕ-Вологда, что может быть объяснено бóльшим размером выборки ЭССЕ-Иваново и указывать на роль размера выборки при репликации результатов GWAS. Тем не менее, реплицированные ВНП с большой надежностью могут быть включены в шкалы генетического риска гиперлипидемий, разрабатываемые для представителей российской популяции.
Обращает на себя внимание, что в данном исследовании удалось подтвердить отмеченную Usoltsev D, et al. (2023) [7] ассоциацию ХС ЛНП с ВНП rs7412 гена APOE. Данный ВНП является известным несинонимичным ВНП, ассоциированным с ХС ЛНП, в частности, в европейской выборке Willer CJ, et al. (2013) [20], а также в российской выборке из центральной России [21]. Для другого ВНП (rs4970834 гена CELSR2) с сильной ассоциацией с ХС ЛНП, заявленной Usoltsev D, et al. (2023) [7], при исследовании выборки ЭССЕ-Иваново ассоциацию подтвердить не удалось. Причиной может служить относительно небольшой размер этой выборки, т.к. в других источниках данный ВНП был отмечен как значимый [22].
Стоит отметить, что все значимые ВНП Usoltsev D, et al. (2023) [7], найденные в выборках ЭССЕ-Вологда и ЭССЕ-Иваново для ХС ЛВП, находятся в гене CETP, и прошли репликацию. Известно, что ген CETP регулирует содержание ХС ЛВП в крови [23]. В частности, была подтверждена ассоциация ВНП rs708272 с уровнем ХС ЛВП. Известно, что данный вариант ассоциирован с более высоким уровнем ХС ЛВП у жителей Западной Сибири европеоидного происхождения [24] Ассоциации ХС ЛВП с вариантами rs34145065 и rs34620476 ранее были описаны в литературе [25], для представителей российской популяции впервые были показаны в данной работе. Единственный значимый ВНП для logТГ из исследования Usoltsev D, et al. (2023) [7] rs964184, который находится в 3`-нетранслируемом участке гена ZPR1, был идентифицирован и реплицирован в обеих выборках. Считается, что rs964184 ассоциирован с уровнем logТГ, т.к. находится около кластера генов APOA5-A4-C3-A1 [26].
Для общего ХС только один ВНП был реплицирован в обеих выборках — rs7412 в гене APOE. Остальные найденные в обеих выборках ВНП (rs1160983, rs4970834, rs1367117), для которых ранее была показана ассоциация с общим ХС [20][22][27], не были реплицированы как минимум в одной из исследуемых выборок. Такой результат, вероятнее всего, объясним относительно небольшим размером выборок ЭССЕ-Вологда и ЭССЕ-Иваново.
Ограничения исследования. Данное исследование имеет ряд ограничений. В первую очередь, это относительно небольшие размеры исследуемых выборок как в случае исходного GWAS (4145 участников), так и для выборок ЭССЕ-Иваново (1675 участников) и ЭССЕ-Вологда (689 участников). Второе важное ограничение данного исследования — ограниченность участков секвенирования выборок ЭССЕ-Вологда и ЭССЕ-Иваново. Панель, использованная для участников выборки ЭССЕ-Вологда, включала 217 генов и дополнительно 18489 ВНП. В случае выборки ЭССЕ-Иваново, панель для секвенирования включала 242 гена и 2042 ВНП.
Было также показано, что показатели липидов статистически значимо различаются в выборках ЭССЕ-Вологда и ЭССЕ-Иваново, что может быть объяснено различиями в половозрастной структуре исследуемых выборок.
Заключение
Поиск ассоциаций позволил идентифицировать ВНП, статистически значимая ассоциация с липидными показателями для которых ранее уже продемонстрирована на выборке лиц, проживающих на территории России. Несмотря на ограниченный объем репликационной выборки, при репликации результатов ранее проведенного GWAS обнаружена высокая степень совпадения как статистической значимости, так и силы эффекта (β-оценки) ВНП, значимо ассоциированных с липидными показателями. Таким образом, полученные на различных выборках представителей российской популяции результаты GWAS для липидных показателей согласуются между собой.
Отношения и деятельность. Работа была поддержана государственным заданием № 121021700364-1 "Разработка интегрированных систем прогнозирования в персонализированной медицине".
1. Hail Team. Hail 0.2.105-acd89e80c345. https://github.com/hail-is/hail/commit/acd89e80c345.
2. https://www.python.org/.
Список литературы
1. Vaduganathan M, Mensah GA, Turco JV, et al. The Global Burden of Cardiovascular Diseases and Risk: A Compass for Future Health. J Am Coll Cardiol. 2022;80(25):2361-71. doi:10.1016/j.jacc.2022.11.005.
2. Ежов М. В., Кухарчук В. В., Сергиенко И. В. и др. Нарушения липидного обмена. Клинические рекомендации 2023. Российский кардиологический журнал. 2023;28(5):5471. doi:10.15829/1560-4071-2023-5471.
3. Choi SW, Mak TS, O'Reilly PF. Tutorial: a guide to performing polygenic risk score analyses. Nat Protoc. 2020;15(9):2759-72. doi:10.1038/s41596-020-0353-1.
4. O'Sullivan JW, Raghavan S, Marquez-Luna C, et al. Polygenic Risk Scores for Cardiovascular Disease: A Scientific Statement from the American Heart Association. Circulation. 2022;146(8):e93118. doi:10.1161/CIR.0000000000001077.
5. Graham SE, Clarke SL, Wu KH, et al. The power of genetic diversity in genome-wide association studies of lipids. Nature. 2021;600(7890):675-9. doi:10.1038/s41586-021-04064-3.
6. Klarin D, Damrauer SM, Cho K, et al. Genetics of blood lipids among ~300,000 multi-ethnic participants of the Million Veteran Program. Nat Genet. 2018;50(11):1514-23. doi:10.1038/s41588018-0222-9.
7. Usoltsev D, Kolosov N, Rotar O, et al. Understanding complex trait susceptibilities and ethnical diversity in a sample of 4,145 russians through analysis of clinical and genetic data. bioRxiv. 2023;23.534000. doi:10.1101/2023.03.23.534000.
8. Marigorta UM, Rodríguez JA, Gibson G, et al. Replicability and Prediction: Lessons and Challenges from GWAS. Trends Genet. 2018;34(7):504-17. doi:10.1016/j.tig.2018.03.005.
9. Бойцов С.А., Чазов Е.И., Шляхто Е.В. и др. Эпидемиология сердечно-сосудистых заболеваний в различных регионах России (ЭССЕ-РФ). Обоснование и дизайн исследований. Профилактическая медицина. 2013;16(6):25-34.
10. Ramensky VE, Ershova AI, Zaicenoka M, et al. Targeted sequencing of 242 clinically important genes in the Russian population from the ivanovo region. Front Genet. 2021;12:709419. doi:10.3389/fgene.2021.709419.
11. Покровская М. С., Борисова А. Л., Метельская В. А. и др. Роль биобанкирования в организации крупномасштабных эпидемиологических исследований. Кардиоваскулярная терапия и профилактика. 2021;20(5):2958. doi:10.15829/1728-88002021-2958.
12. Копылова О. В., Ершова А. И., Покровская М. С. и др. Популяционно-нозологический исследовательский биобанк "НМИЦ ТПМ": анализ коллекций биообразцов, принципы сбора и хранения информации. Кардиоваскулярная терапия и профилактика. 2021;20(8):3119. doi:10.15829/1728-8800-20213119.
13. Poplin R, Ruano-Rubio V, DePristo MA, et al. Scaling accurate genetic variant discovery to tens of thousands of samples. bioRxiv. 2017. doi:10.1101/201178.
14. McLaren W, Gil L, Hunt SE, et al. The Ensembl Variant Effect Predictor. Genome Biol. 2016;17(1):122. doi:10.1186/s13059016-0974-4.
15. Sherry ST, Ward MH, Kholodov M, et al. dbSNP: the NCBI database of genetic variation. Nucleic Acids Res. 2001;29(1):308-11. doi:10.1093/nar/29.1.308.
16. Hinrichs AS, Karolchik D, Baertsch R et al. The UCSC Genome Browser Database: update 2006. Nucleic Acids Res. 2006;34(90001):D590-8. doi:10.1093/nar/gkj144.
17. Virtanen P, Gommers R, Oliphant TE, et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nat Methods. 2020;17(3):261-72. doi:10.1038/s41592-019-0686-2.
18. Gonzalez S, Gupta J, Villa E, et al. Replication of genome‐wide association study (GWAS) susceptibility loci in a Latino bipolar disorder cohort. Bipolar Dis. 2016;18(6):520-7. doi:10.1111/bdi.12438.
19. Selvaraj MS, Li X, Li Z, et al. Whole genome sequence analysis of blood lipid levels in >66,000 individuals. Nat Commun. 2022;13(1). doi:10.1038/s41467-022-33510-7.
20. Willer CJ, Schmidt EM, Sengupta S, et al. Discovery and refinement of loci associated with lipid levels. Nat Genet. 2013; 45(11):1274-83. doi:10.1038/ng.2797.
21. Чурилин М. И., Кононов С. И., Лунева Ю. В. и др. Полиморфные варианты гена аполипопротеина E: связь с риском развития ишемической болезни сердца и эффективностью гиполипидемической терапии розувастатином. Кардиоваскулярная терапия и профилактика. 2020;19(1):17-23. doi:10.15829/1728-8800-2020-1-2297.
22. Richardson TG, Sanderson E, Palmer TM, et al. Evaluating the relationship between circulating lipoprotein lipids and apolipoproteins with risk of coronary heart disease: A multivariable Mendelian randomisation analysis. PLoS Med. 2020; 17(3). doi:10.1371/journal.pmed.1003062.
23. Бушуева О. Ю., Стецкая Т. А., Корогодина Т. В. и др. Исследование взаимосвязи полиморфизмов HindIII гена LPL и Taq1b гена CETP с риском развития атеротромботического инсульта у жителей Центральной России. Терапевтический архив. 2015;87(8):86-91. doi:10.17116/terarkh201587886-91.
24. Шахтшнейдер Е. В., Куликов И. В., Максимов В.Н. и др. Полиморфизм гена CETP в европеоидной популяции западной Сибири и группах контрастных по уровню ОХС. (Бюллетень экспериментальной биологии и медицины. 2014;157(3):364-7. doi:10.1007/s10517-014-2567-0.
25. Prins BP, Kuchenbaecker KB, Bao Y, et al. Genome-wide analysis of health-related biomarkers in the UK Household Longitudinal Study reveals novel associations. Sci Rep. 2017;7(1):11008. doi:10.1038/s41598-017-10812-1.
26. Van de Woestijne AP, van der Graaf Y, de Bakker PIW, et al. Rs964184 (APOA5-A4-C3-A1) Is Related to Elevated Plasma Triglyceride Levels, but Not to an Increased Risk for Vascular Events in Patients with Clinically Manifest Vascular Disease. Calabresi L, editor. PLoS ONE. 2014;9(6):e101082. doi:10.1371/journal.pone.0101082.
27. Barton AR, Sherman MA, Mukamel RE, et al. Whole-exome imputation within UK Biobank powers rare coding variant association and fine-mapping analyses. Nat Gen. 2021;53(8):1260-9. doi:10.1038/s41588-021-00892-1.
Об авторах
М. ЗайченокаЛатвия
Аспирант.
Московская область, Долгопрудный
А. И. Ершова
Россия
д.м.н., руководитель лаборатории клиномики, зам. директора по фундаментальной науке.
Москва
А. В. Киселева
Россия
к.б.н., руководитель, в.н.с.
Москва
Е. А. Сотникова
Россия
с.н.с.
Москва
Ю. В. Вяткин
Россия
с.н.с., программист, Институт перспективных исследований проблем искусственного интеллекта и интеллектуальных систем.
Москва
А. А. Жарикова
Россия
к.б.н., в.н.с., старший преподаватель, факультет биоинженерии и биоинформатики.
Москва
М. С. Покровская
Россия
к.б.н., в.н.с., руководитель лаборатории "Банк биологического материала" Института персонализированной терапии и профилактики, в.н.с.
Москва
С. А. Шальнова
Россия
д.м.н., профессор, руководитель отдела эпидемиологии хронических неинфекционных заболеваний.
Москва
В. Е. Раменский
Россия
руководитель лаборатории геномной и медицинской биоинформатики, в.н.с., доцент, руководитель научной группы "ИИ в биоинформатике и медицине", Институт перспективных исследований проблем искусственного интеллекта и интеллектуальных систем, факультет биоинженерии и биоинформатики.
Москва
А. Н. Мешков
Россия
д.м.н., руководитель Института персонализированной терапии и профилактики.
Москва
О. М. Драпкина
Россия
д.м.н., профессор, академик РАН, директор.
Москва
Дополнительные файлы
Что известно о предмете исследования?
- Недавно в работе Usoltsev D, et al. (2023) опубликованы результаты первого полногеномного поиска ассоциаций для липидных показателей, проведенного на выборке из представителей российской популяции из трех регионов РФ: Санкт Петербурга, Самарской и Оренбургской областей.
- Результаты полногеномных исследований ассоциаций нуждаются в репликации на независимых выборках.
Что добавляют результаты исследования?
- Проведена репликация результатов полногеномного поиска ассоциаций для показателей липидного профиля на двух выборках представителей российской популяции из Вологодской и Ивановской областей.
- Результаты поиска ассоциаций с липидными показателями, полученные на разных выборках представителей российской популяции, согласуются между собой.
Рецензия
Для цитирования:
Зайченока М., Ершова А.И., Киселева А.В., Сотникова Е.А., Вяткин Ю.В., Жарикова А.А., Покровская М.С., Шальнова С.А., Раменский В.Е., Мешков А.Н., Драпкина О.М. Поиск и репликация ассоциаций вариантов генома с уровнями липидов в выборке из представителей российской популяции. Кардиоваскулярная терапия и профилактика. 2023;22(12):3871. https://doi.org/10.15829/1728-8800-2023-3871. EDN: GTFBBS
For citation:
Zaichenoka M., Ershova A.I., Kiseleva A.V., Sotnikova E.A., Vyatkin Yu.V., Zharikova А.A., Pokrovskaya M.S., Shalnova S.A., Ramensky V.E., Meshkov A.N., Drapkina O.M. Search and replication of associations of genome variants with lipid levels in a Russian sample. Cardiovascular Therapy and Prevention. 2023;22(12):3871. (In Russ.) https://doi.org/10.15829/1728-8800-2023-3871. EDN: GTFBBS
JATS XML

























































